3.4. Các mô hình gắn kết phân tử docking của các dẫn xuất benzimidazole và indole
Kết quả re-docking các ligand đồng kết tinh CPT vào tinh thể phức hợp TopI- DNA được trình bày ở bảng 3.6 và hình 3.4.
Thụ thể | Tên Protein | Tên ligand đồng kết tinh | Docking với | ĐIỂM SỐ DOCKING (kJ.mol-1) | RMSD (Å) |
Phức hợp protein TopI- DNA của người với CPT và cộng hóa trị với một chuỗi DNA xoắn kép. | Ligand đồng kết tách ra từ tinh thể | -25,89 | 0,6602 | ||
Ligand đồng kết tinh tách ra từ tinh thể có xử lý theo các bước đã trình bày | -29,50 | 1,3540 | |||
1T8I [101] | CPT | ||||
Ligand được chuẩn bị ngay từ đầu và có xử lý theo các bước đã trình bày | -27,61 | 1,5686 |
Có thể bạn quan tâm!
-
![[(4-Methoxy-Phenyl)-Pyrrolidin-1-Yl-Methyl]-1H-Indole-2- Carboxylic Acid (Cpm – Chất Mới)](data:image/svg+xml,%3Csvg%20xmlns=%22http://www.w3.org/2000/svg%22%20viewBox=%220%200%2075%2075%22%3E%3C/svg%3E) [(4-Methoxy-Phenyl)-Pyrrolidin-1-Yl-Methyl]-1H-Indole-2- Carboxylic Acid (Cpm – Chất Mới)
[(4-Methoxy-Phenyl)-Pyrrolidin-1-Yl-Methyl]-1H-Indole-2- Carboxylic Acid (Cpm – Chất Mới) -
![Bromo-3-[(4-Methoxy-Phenyl)-Pyrrolidin-1-Yl-Methyl]-1H-Indole (Bpm)](data:image/svg+xml,%3Csvg%20xmlns=%22http://www.w3.org/2000/svg%22%20viewBox=%220%200%2075%2075%22%3E%3C/svg%3E) Bromo-3-[(4-Methoxy-Phenyl)-Pyrrolidin-1-Yl-Methyl]-1H-Indole (Bpm)
Bromo-3-[(4-Methoxy-Phenyl)-Pyrrolidin-1-Yl-Methyl]-1H-Indole (Bpm) -
 Mối Quan Hệ Giữa Cấu Trúc Của Các Dẫn Xuất Benzimidazole Tổng Hợp Và Hoạt Tính Ức Chế Tế Bào Ung Thư
Mối Quan Hệ Giữa Cấu Trúc Của Các Dẫn Xuất Benzimidazole Tổng Hợp Và Hoạt Tính Ức Chế Tế Bào Ung Thư -
 Nghiên cứu tổng hợp và đánh giá hoạt tính gây độc tế bào các dẫn xuất của benzimidazole và indole - 21
Nghiên cứu tổng hợp và đánh giá hoạt tính gây độc tế bào các dẫn xuất của benzimidazole và indole - 21 -
 Nghiên cứu tổng hợp và đánh giá hoạt tính gây độc tế bào các dẫn xuất của benzimidazole và indole - 22
Nghiên cứu tổng hợp và đánh giá hoạt tính gây độc tế bào các dẫn xuất của benzimidazole và indole - 22 -
 Nghiên cứu tổng hợp và đánh giá hoạt tính gây độc tế bào các dẫn xuất của benzimidazole và indole - 23
Nghiên cứu tổng hợp và đánh giá hoạt tính gây độc tế bào các dẫn xuất của benzimidazole và indole - 23
Xem toàn bộ 186 trang tài liệu này.
Bảng 3.6. Kết quả re-docking với phức hợp TopI-DNA (PDB ID: 1T8I)
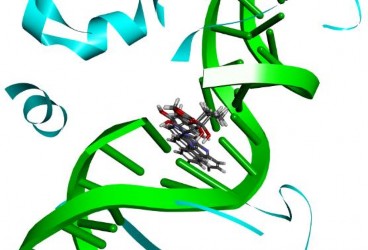
Hình 3.4. Xếp chồng các cấu dạng của ligand đồng kết tinh CPT vào tinh thể của phức hợp TopI-DNA (PDB: 1T8I). Chuỗi kép DNA và TopI được biểu diễn bằng các dải băng màu xanh lá và xanh lam
3.4.1. Mô hình gắn kết phân tử docking của các dẫn xuất benzimidazole có hoạt tính ức chế tế bào tốt nhất trên phức hợp TopI-DNA
Mô hình mô tả phân tử docking được xây dựng nhằm đề xuất cơ chế gây độc tế bào của các dẫn xuất benzimidazole có hoạt tính kháng ung thư tốt nhất 5MM, 53H và 3BO.
Bảng 3.7. Kết quả docking của ba dẫn xuất benzimidazole 5MM, 53H và 3BO và camptothecin trên phức hợp TopI-DNA (PDB ID: 1T8I)
IC50 ± SD (µM) ĐIỂM SỐ
DẪN XUẤT
(kJ.mol-1) | ||||
5MM | > 100 | 10,82 ± 0,35 | > 100 | -25,85 |
53H | 14,24 ± 0,33 | 14,90 ± 0,42 | 17,52 ± 0,28 | -22,46 |
3BO | > 100 | 19,79 ± 0,83 | 59,73 ± 1,08 | (*) |
CPT | 0,57 ± 0,06 | 1,35 ± 0,04 | 2,50 ± 0,11 | -29,50 |
A549 MDA-MB-231 PC3
DOCKING
(*) không docking được vào khoang gắn kết
Kết quả docking của các dẫn xuất benzimidazole 5MM, 53H, 3BO và CPT được biểu diễn ở bảng 3.7, kết quả cho thấy ligand 5MM, 53H docking thành công vào khoang gắn kết CPT của phức hợp protein (hình 3.5 đến 3.7), ligand có điểm số docking cao nhất là CPT (-29,50 kJ.mol-1), kế tiếp đó lần lượt là 5MM (-25,85 kJ.mol- 1), 53H (-22,46 kJ.mol-1). Dẫn xuất 3BO docking không thành công vào khoang gắn
kết CPT của phức hợp TopI-DNA. Nguyên nhân có thể do cấu hình của 3BO và thuật toán docking của FlexX. Thuật toán docking gồm 3 bước: lựa chọn mảnh cơ sở, định vị mảnh cơ sở và xây dựng cấu dạng gắn kết của ligand, khi bất kì bước nào của thuật toán không thể thỏa, đều dẫn đến việc ligand docking không thành công vào protein. Ví dụ ở giai đoạn định vị mảnh cơ sở dựa theo thuật toán tam giác phân, thuật toán sẽ dừng việc định vị khi có ít nhất 3 vị trí tương tác giữa phức hợp protein TopI-DNA và mảnh cơ sở của 3BO được tìm thấy, khi không thỏa điều kiện này, quá trình docking sẽ kết thúc và không ghi nhận được kết quả [133].
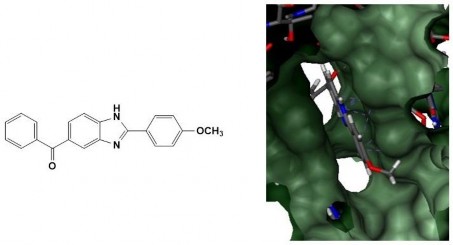
Hình 3.5. Dẫn xuất 5MM tại khoang gắn kết CPT của phức hợp TopI-DNA
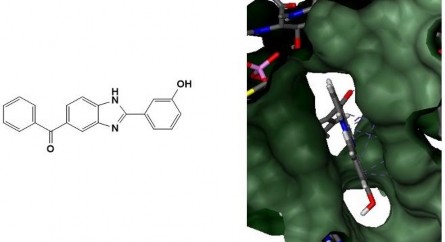
Hình 3.6. Dẫn xuất 53H tại khoang gắn kết CPT của phức hợp TopI-DNA
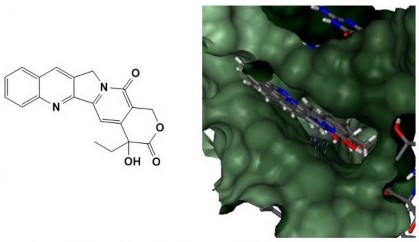
Hình 3.7. Camptothecin tại khoang gắn kết của phức hợp TopI-DNA
Ngoài ra, 5MM và 53H chèn vào vị trí gắn kết của phức hợp TopI-DNA, tương tự như cách thức gắn kết của CPT trong khoang gắn kết của phức hợp này (Intercalation), được biểu diễn ở hình 3.8 (A, C, và E). Tương tác amino acid của 5MM, 53H và CPT với khoang gắn kết CPT của phức hợp TopI-DNA (PDB: 1T8I) được biểu diễn ở hình 3.8 (B, D, và F). Kết quả cho thấy, CPT cũng như 5MM và 53H đều có tương tác hydro với amino acid Asp533 bằng liên kết hydro, đây cũng là amino acid quan trọng đặc trưng cho độ nhạy của phức hợp TopI-DNA với CPT [101, 134]. Ngoài ra, 5MM và 53H không tạo liên kết hydro với amino acid Asn722 và đặc biệt là thiếu tương tác với chuỗi DNA thông qua cặp base dt10, do đó không tạo nên được phức hợp bậc 3 CPT-topI-DNA như CPT [101, 135], dẫn đến hoạt tính sinh học của 5MM và 53H kém hơn so với CPT. Bên cạnh đó, các ligand còn tạo được tương tác kỵ nước với với các amino acid và các base khác trong khoang gắn kết như: với CPT (base: da113 và dc112; amino acid: Tgp11 và Thr718), 5MM (base: da113, dt10 và dg12; amino acid: Tgp11 và Arg364) và 53H (base: dt10, da113 và dg12; amino acid: Arg364 và Tgp11).
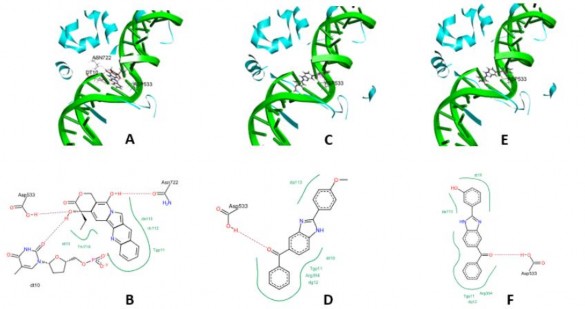
Chuỗi kép DNA và TopI được biểu diễn bằng các dải băng màu xanh lá và xanh lam; liên kết hydro được thể hiện bằng nét đứt, liên kết kỵ nước được vẽ bằng đường cong màu xanh lá
Hình 3.8. Mô hình 3D và 2D của CPT (A và B), 5MM (C và D), và 53H (E và F) chèn vào khoang gắn kết của phức hợp TopI-DNA
3.4.2. Mô hình gắn kết phân tử docking của các dẫn xuất indole có hoạt tính ức chế tế bào tốt nhất trên phức hợp TopI-DNA
Mô hình mô tả phân tử docking được xây dựng nhằm đề xuất cơ chế gây độc tế bào của các dẫn xuất indole có hoạt tính kháng ung thư tốt nhất BPM, IPM, và PPM. Điểm số docking của ba dẫn xuất indole này và CPT được thể hiện ở bảng 3.8. Bảng 3.8. Kết quả docking của 3 dẫn xuất indole tốt nhất và camptothecin trên enzyme 1T8I
DẪN XUẤT
IC50 ± SD (µM) ĐIỂM SỐ
DOCKING
(kJ.mol-1) | ||||
BPM | 8,83 ± 0,43 | 9,24 ± 0,15 | 9,90 ± 0,28 | -22,69 |
IPM | 19,25 ± 0,56 | 19,71 ± 0,24 | 21,11 ± 0,34 | -21,33 |
PPM | 29,37 ± 0,47 | 38,72 ± 0,98 | > 100 | -10,97 |
CPT | 0,57 ± 0,06 | 1,35 ± 0,04 | 2,50 ± 0,11 | -29,50 |
A549 MDA-MB-231 PC3
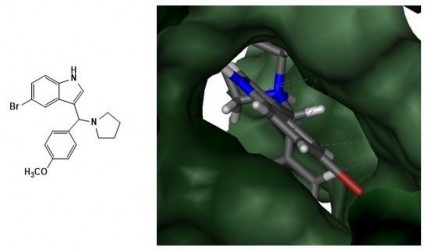
Hình 3.9. Dẫn xuất BPM tại khoang gắn kết CPT của phức hợp TopI-DNA
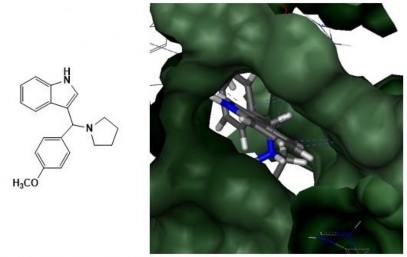
Hình 3.10. Dẫn xuất IPM tại khoang gắn kết CPT của phức hợp TopI-DNA
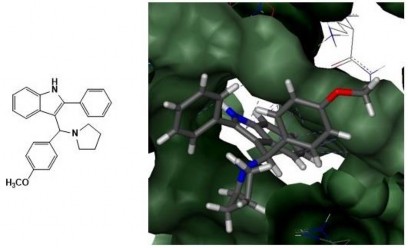
Hình 3.11. Dẫn xuất PPM tại khoang gắn kết CPT của phức hợp TopI-DNA Kết quả cho thấy cả 3 dẫn xuất indole BPM, IPM và PPM docking thành công
vào khoang gắn kết CPT của phức hợp TopI-DNA (hình 3.9 đến 3.11), ligand có điểm số docking cao nhất là CPT (-29,5089 kJ.mol-1), kế tiếp đó lần lượt là BPM (-22,69 kJ.mol-1), IPM (-21,33 kJ.mol-1) và PPM (-10,97 kJ.mol-1).
BPM, IPM, PPM chèn vào vị trí gắn kết của phức hợp TopI-DNA, tương tự như cách thức gắn kết của CPT trong khoang gắn kết của phức hợp này (Intercalation), được biểu diễn ở hình 3.12 (A, C, và E). Tương tác của BPM, IPM, và PPM với các amino acid của phức hợp TopI-DNA được biểu diễn ở hình 3.12 (B, D, và F). Kết quả cho thấy CPT cũng như BPM đều có tương tác với amino acid Asp533 (amino acid quan trọng đặc trưng cho độ nhạy của enzyme TopI với CPT [101, 134]) và Asn722 bằng liên kết hydro. Hai dẫn xuất IPM và PPM chỉ tạo liên kết hydro với amino acid Asn722, do đó hoạt tính ức chế tế bào ung thư của IPM và PPM kém hơn hẳn so với BPM. Ngoài ra, BPM, IPM và PPM đều thiếu tương tác hydro với chuỗi DNA thông qua cặp base dt10, do đó không tạo nên được phức hợp bậc 3 CPT-topI-DNA như CPT [101, 135], dẫn đến hoạt tính sinh học in vitro của cả 3 dẫn xuất đều kém hơn so với CPT. Bên cạnh đó, các ligand còn tạo được tương tác kỵ nước với với các amino acid khác trong khoang gắn kết như: CPT (base: da113 và dc112; amino acid: Tgp11 và Thr718), BPM (base: dg12, da113 và dt10; amino acid: Lys532, Thr718 và Arg364) and IPM (base: dg12, da113 và dt10; amino acid: His632, Thr718 và Tgp11) và PPM (base: dt10 và da113; amino acid: Tgp11 và Asn722).
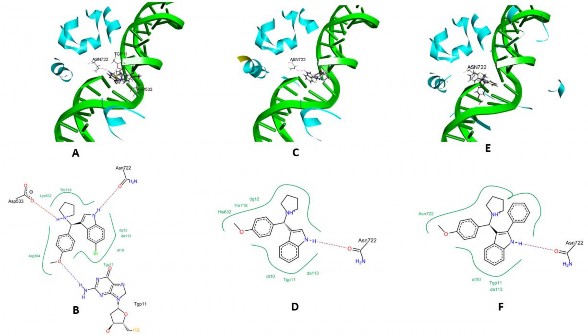
Chuỗi kép DNA và TopI được biểu diễn bằng các dải băng màu xanh lá và xanh lam; liên kết hydro được thể hiện bằng nét đứt, liên kết kỵ nước được vẽ bằng đường cong màu xanh lá
Hình 3.12. Mô hình 3D và 2D của BPM (A và B), IPM (C và D), và PPM (E và F) chèn vào khoang gắn kết của phức hợp TopI-DNA
Từ các dữ liệu mô hình gắn kết phân tử với hoạt tính sinh học in vitro của các dẫn xuất benzimidazole (5MM, 53H) và indole (BPM, IPM, PPM) trên đối tượng tác động là phức hợp TopI-DNA(PDB: 1T8I) ta có thể thấy, các dẫn xuất này có cách thức gắn kết vào phức hợp TopI-DNA theo kiểu xen kẽ, chèn vào (Intercalation) vị trí khoang hoạt động của phức hợp TopI-DNA, tương tự như cách thức gắn kết của CPT. Cách thức gắn kết này ngăn không cho tương tác giữa TopI và DNA xảy ra, từ đó dẫn tới TopI không thực hiện được chức năng tháo xoắn cho chuỗi DNA, ảnh hưởng tới quá trình phát triển của tế bào. Ngoài ra, các dẫn xuất có hoạt tính ức chế tế bào ung thư cao nhất BPM, 5MM và 53H, giống như ligand CPT, đều có chung đặc điểm tạo được liên kết hydro với amino acid Asp533 – đặc trưng cho độ nhạy của enzyme trong khoang gắn kết CPT và đã được xác nhận trong nghiên cứu của Staker và cộng sự [101]. Hai dẫn xuất indole còn lại IPM và PPM có hoạt tính kém hơn và đồng thời cũng không tạo được liên kết hydro với amino acid quan trọng này. Hơn nữa, BPM tạo được liên kết hydro cùng lúc với amino acid Asp533 và Asn722 (tương tự như CPT) nên BPM có hoạt tính ức chế trên dòng tế bào MDA-MB-231 cao nhất

![[(4-Methoxy-Phenyl)-Pyrrolidin-1-Yl-Methyl]-1H-Indole-2- Carboxylic Acid (Cpm – Chất Mới)](https://tailieuthamkhao.com/uploads/2022/09/02/nghien-cuu-tong-hop-va-danh-gia-hoat-tinh-gay-doc-te-bao-cac-dan-xuat-17-120x90.jpg)
![Bromo-3-[(4-Methoxy-Phenyl)-Pyrrolidin-1-Yl-Methyl]-1H-Indole (Bpm)](https://tailieuthamkhao.com/uploads/2022/09/02/nghien-cuu-tong-hop-va-danh-gia-hoat-tinh-gay-doc-te-bao-cac-dan-xuat-18-120x90.jpg)