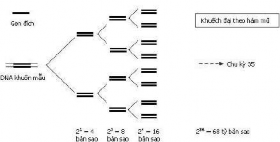
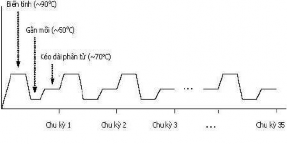
(a) Sơ đồ phản ứng chuỗi polymerase (b) Các chu kỳ của phản ứng chuỗi
polymerase
Hình 1.3. Sơ đồ và các chu kỳ của phản ứng chuỗi polymerase [15]
1.4.2.2. Các yếu tố ảnh hưởng đến phản ứng PCR
- ADN mẫu: Phản ứng khuếch đại tối ưu xảy ra trên ADN thật tinh sạch. Nhiều kỹ thuật chẩn đoán bằng PCR vẫn đạt kết quả tốt với ADN thu nhận được trực tiếp từ dịch chiết tế bào. Lượng ADN mẫu sử dụng cũng có xu hướng giảm (1 µg xuống còn 100 ng) với việc sử dụng các DNA polymerase có hiệu quả cao [6].
- DNA polymerase: Là một loại enzym chịu nhiệt giúp cho sự tổng hợp sợi ADN mới bằng cách bổ sung các nucleotid mới và trình tự đích. Taq DNA polymerase là enzym đầu tiên và phổ biến nhất được tách chiết từ vi khuẩn sống ở các suối nước nóng Thermus aquaticus. Enzym này không bị phá vỡ ở nhiệt độ biến tính. Ngày nay, nhiều polymerase chịu nhiệt khác được đưa ra thị trường với nhiều chức năng chuyên biệt và hoàn thiện hơn [6].
- Đoạn mồi (primer): Primer là đoạn ngắn ADN đơn mà nó có chứa trình tự đích. Enzym polymerase bắt đầu tổng hợp sợi ADN mới từ đầu 3’ của primer. Primer là chìa khóa quan trọng cho sự thành công hay thất bại của một thí nghiệm PCR. Nếu primer được thiết kế một cách chính xác thì thí nghiệm sẽ mang lại kết quả về sự khuếch đại của một mảnh ADN đơn [6].
Có thể bạn quan tâm!
-
 Nghiên cứu nâng cấp tiêu chuẩn dược liệu rễ Ba kích Radix Morindae officinalis của Việt Nam - 2
Nghiên cứu nâng cấp tiêu chuẩn dược liệu rễ Ba kích Radix Morindae officinalis của Việt Nam - 2 -
 Tóm Tắt Chuyên Luận Dược Liệu Rễ Ba Kích Trong Dược Điển
Tóm Tắt Chuyên Luận Dược Liệu Rễ Ba Kích Trong Dược Điển -
 Tình Hình Thiết Lập Chất Chuẩn Monotropein Và Nystose
Tình Hình Thiết Lập Chất Chuẩn Monotropein Và Nystose -
 Thẩm Định Phương Pháp Định Tính, Định Lượng Monotropein Và Nystose
Thẩm Định Phương Pháp Định Tính, Định Lượng Monotropein Và Nystose -
![Nội Dung Thẩm Định Phương Pháp Định Lượng Nystose [52]](data:image/svg+xml,%3Csvg%20xmlns=%22http://www.w3.org/2000/svg%22%20viewBox=%220%200%2075%2075%22%3E%3C/svg%3E) Nội Dung Thẩm Định Phương Pháp Định Lượng Nystose [52]
Nội Dung Thẩm Định Phương Pháp Định Lượng Nystose [52] -
 Xây Dựng Tiêu Chuẩn Chất Lượng Chất Chuẩn Monotropein Và Nystose
Xây Dựng Tiêu Chuẩn Chất Lượng Chất Chuẩn Monotropein Và Nystose
Xem toàn bộ 226 trang tài liệu này.
- Nồng độ dNTP (các deoxynucleotid triphotphat): Nồng độ dNTP thường được sử dụng là 20 – 200 μM. Nồng độ cao hơn dễ dẫn đến sự khuếch đại “ký sinh". Bên cạnh đó, sự cân bằng trong thành phần các dNTP cũng ảnh hưởng đến phản ứng PCR. Sự mất cân bằng trong thành phần các dNTP sẽ làm tăng các lỗi sao chép của DNA polymerase [6].
- Nồng độ MgCl2: Nồng độ MgCl2 cũng là một nhân tố ảnh hưởng mạnh đến phản ứng PCR. Mg2+ rất cần cho quá trình liên kết các dNTP, xúc tác cho enzym Taq polymerase, làm tăng Tm của ADN mạch kép. Mg2+ là Co – factor của Taq polymerase
nên nếu lượng Mg2+ quá thấp thì Taq polymerase sẽ không thể hoạt động bình thường trong giai đoạn kéo dài, hạn chế quá trình kéo dài. Ngược lại nồng độ Mg2+ cao sẽ giúp ổn định dây đôi ADN và ngăn ngừa sự biến tính hoàn toàn (do sự mở dây đôi ADN) của sản phẩm trong mỗi chu kỳ, làm cho sản phẩm PCR ít đi; đồng thời, có thể làm cho hiện tượng bắt cặp giả xảy ra ổn định hơn và cho ra những sản phẩm mong muốn với số lượng quá lớn nhưng mức độ chuyên biệt thấp [6].
- Dung dịch đệm: Dung dịch đệm có nồng độ cao được sử dụng để tăng cường hiệu quả cho phản ứng PCR [6].
- Số lượng chu kỳ của phản ứng PCR: Trong thực tế số chu kỳ cho một phản ứng PCR không nên vượt quá 40. Sở dĩ như vậy vì phản ứng PCR diễn tiến qua hai giai đoạn [6]:
+ Giai đoạn đầu: Số lượng bản sao tăng lên theo cấp số nhân, tỉ lệ với lượng mẫu ban đầu.
+ Giai đoạn sau: Hiệu quả khuếch đại giảm hẳn do sự phân hủy và cạn kiệt các thành phần của phản ứng, sự xuất hiện các sản phẩm phụ ức chế phản ứng, hoặc các bản sao vừa được tổng hợp không kết hợp với mồi mà bắt cặp với nhau.
1.4.3. Các trình tự gen thường dùng cho thực vật
Sau nhiều nghiên cứu so sánh và đánh giá, hiện nay 3 gen của lục lạp (rbcL, matK, trnH-psbA) và 1 vùng gen của nhiễm sắc thể (ITS bao gồm ITS 1, ITS 2) đang được dùng song song hoặc phối hợp [54]. Ngoài ra, để đạt được các mục đích phân tích khác nhau người ta có thể chọn các đoạn gene đích khác để so sánh như 5.8S rDNA, 18S rDNA, 26S rDNA, 5S rDNA spacer, trên nhân hoặc trnL-trnF, trnL, atpB-rbcL, matK trên lục lạp... hoặc phối hợp nhiều gen cùng lúc để đạt độ tin cậy cao hơn [32], [87].
1.4.3.1. Trình tự gen nhân
Các trình tự mã vạch được nhân bản từ ADN hệ gen của bố mẹ dự kiến sẽ cung cấp nhiều hơn thông tin về các loài cần xác định. Tuy nhiên khó khăn trong việc khuếch đại PCR từ gen nhân vì gen nhân chủ yếu là đơn gen hoặc có bản sao gen thấp, đặc biệt từ sự suy thoái về chất lượng gen ADN và khả năng phân biệt dưới loài do bảo toàn gen chức năng có thể chính là lý do tại sao hạn chế số lượng các gen nhân được thử nghiệm trong xác định loài bằng phương pháp mã vạch ADN [50], [83].
1.4.3.2. Vùng gen mã hóa ribosome
* Trình tự ITS
Các trình tự ITS (internal transcribed spacer) là chuỗi trình tự ADN nằm xen kẽ được sao chép giữa các gen mã hóa ribosome ARN (rRNA) 18S–5.8S–26S, đây là một nhóm trình tự thuộc loại cụm đa gen (multigene families). Trình tự này được dùng phổ biến nhất trong việc phân loại thực vật và nấm vì chúng có các ưu điểm:
- Di truyền theo cả 2 phía cá thể cha mẹ (biparental inheritance) so với tình trạng di truyền theo 1 phía cá thể mẹ như ty thể và lạp thể.
- Dễ nhân bản vì có nhiều mồi dùng chung (universal primers) cho nhiều ngành sinh vật khác nhau.
- Có nhiều bản sao nên dễ phân lập và nhân bản, kể cả khi phân tích mẫu vật đã
xử lý hoặc lâu năm [25], [70].
- Kích thước ngắn nên dễ dàng giải trình tự.
- Theo nhiều nghiên cứu đã công bố thì chúng được đánh giá là phù hợp để phân loại đến cấp loài.
- Ngoài ra, cụm đa gen này đáp ứng các yêu cầu của một ứng viên làm gen đích cho các phân tích chỉ thị mã vạch vì chúng nằm liền kề những đoạn có tính bảo tồn nghiêm ngặt mã hóa cho rRNA xen kẽ với vùng tiến hóa [31], [70].
Sơ đồ vị trí các vùng ITS trên cụm đa gen 18S–5.8S–26S rRNA được thể hiện tại Hình 1.4.
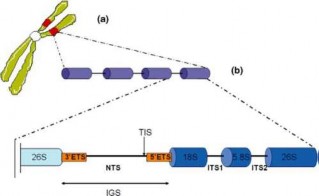
Hình 1.4. Vị trí các vùng ITS trên cụm đa gen 18S–5.8S–26S rRNA [70]
Chiều dài tổng của vùng gen 18S–5.8S–26S rRNA vào khoảng 500-700bp ở
ngành hạt kín và khoảng 1.500-3.500 bp ở ngành hạt trần [70].
1.4.4.3. Các trình tự trên lục lạp
Lục lạp trong các cây trên cạn là thành quả của quá trình nội cộng sinh xảy ra giữa khuẩn lam (Cyanobacteria) với vi sinh vật chủ nhân thật (Eukaryote). Vì sự cộng sinh và đồng tiến hóa ảnh hưởng qua lại giữa thực vật và lục lạp rất chặt chẽ nên hệ gen của lục lạp dần dần thoái biến và lục lạp phụ thuộc vào ký chủ như một bào quan. Bộ gen lục lạp có kích thước trung bình khoảng 145 kbp với khoảng 101- 118 gen khác nhau Trong khi đó, những loài khuẩn lam sống tự nhiên như Nostoc sp. PCC 7120 có 6.4 Mb mã hóa khoảng 6.626 gen và Synechocystis sp. có 3.5 Mb mã hóa khoảng 4.003 gen. Như vậy, bộ gen lục lạp chiếm khoảng dưới 5% bộ gen của khuẩn lam sống tự do tương ứng [30], [71], [75].
Bộ gen của lục lạp (cpDNA) nói riêng và lạp thể (ptDNA) nói chung, được các nhà phân loại học đặc biệt quan tâm là do trật tự sắp xếp các gen gần như giống nhau và sự tiến hóa của nó tương đối ổn định hơn so với bộ gen của nhiễm sắc thể. Ngoài ra, do lục lạp phân chia độc lập và di truyền đơn hệ (mẫu hệ) nên không có sự nhảy gen (transposon) và không có sự tái tổ hợp [71]. Đây là những đặc điểm gần như là hoàn hảo để áp dụng các thuật toán so sánh. Vì vậy, phần lớn các nghiên cứu cây phát sinh loài thực vật dựa trên sinh học phân tử đều dựa trên bộ gen của lục lạp. Tuy nhiên, bộ gen này có nhược điểm là do tiến hóa chậm nên chúng khó cung cấp thông tin phân biệt ở mức độ dưới loài.
Các gen trên cpDNA được chia làm 4 vùng, trong đó có 2 vùng gen lặp lại đảo ngược (inverted repeat - IR) bị chia cách bởi 2 vùng đơn gen lớn (large single copy - LSC) và vùng đơn gen nhỏ (small single copy - SSC). Các gen nằm trong vùng IR thường có độ bảo tồn cao và tiến hóa chậm trong khi đó các gen nằm trong vùng LSC, và SSC tiến hóa nhanh hơn (Hình 1.5).
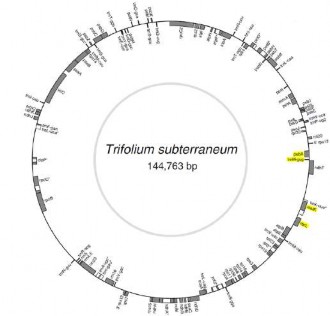
Hình 1.5. Vị trí các gen trên cpDNA của Trifolium subterraneum [88]
* Trình tự rbcL
Gen rbcL trên lục lạp (Rubisco large subunit gen) là gen mã hóa cấu tử protein kích thước lớn của Rubisco. Chiều dài gen thường ổn định ở mức 1.428 bp, 1.431 bp và 1.434 bp và rất hiếm khi xảy ra hiện tượng chèn/mất nucleotid (indels). Gen rbcL không những được dùng phổ biến ở thực vật mà còn dùng trong phân loại dương xỉ, nhất là ở mức độ phân biệt cấp họ trở lên [75]. Phân tích gen rbcL được Chase và cộng sự [32] tiến hành lần đầu vào năm 1993, thực hiện trên 499 loài bao gồm nhiều bộ, họ thực vật phổ biến nhất như Asteridae, Cornaceae, Ericales, Magnoliaceae, Zingiberaceae,... và một số bộ, họ nhỏ thì có thể chỉ có 1 vài đại diện. Việc phân tích rbcL không những áp dụng cho các loài còn đang tồn tại mà còn được áp dụng cho các mẫu vật khảo cổ có niên đại khoảng 17 - 20 triệu năm [75].
Để khuếch đại rbcL người ta có thể dùng mồi chung (universal) hoặc dùng nhiều cặp mồi đặc hiệu khác nhau tùy thuộc vào ngành thực vật như Tảo (Chlorophyta, Rhodophyta), Dương xỉ (Polypodiophyta), hoặc cây có hạt (Angiosperms, Gymnosperms) nhằm nâng cao tính đặc hiệu [75].
* Trình tự matK
Trình tự matK là gen mã hóa một protein maturase được Neuhaus H., Link G. đề xuất năm 1987 [69] nhưng đến năm 1995 mới được Johnson và Soltis dùng vào việc phân loại [53].
Vị trí của matK khá đặc biệt, matK nằm chèn trên vùng intron dài khoảng 2.600 bp của 2 exon là exon 5’ trnK và exon 3’trnK (Hình 1.6) mã hóa cho tRNA vận chuyển lysine (trnK). Các gen kế tiếp trên cpDNA về phía 5’ là rps16 và phía 3’ là psbA có độ ổn định cao nên có thể sẽ làm nguồn thông tin để thiết kế mồi nếu muốn khuếch đại toàn bộ cụm gen trnK và matK. Tổng chiều dài matK là 1.550 bp nhưng trong nghiên cứu của Johnson và Soltis chỉ khảo sát tổng cộng 1078 bp vùng bắt đầu từ 5’.
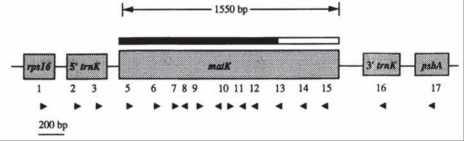
Hình 1.6. Vị trí tương đối của matK so với trnK trên cpDNA [75]
Cho đến nay thì khi nghiên cứu vùng gen trnK người ta chỉ chú trọng đến giải trình tự matK, nhưng việc kết hợp các vùng trình tự xen kẽ có thể có thêm nhiều thông tin hữu ích. Trong nghiên cứu cây hạt kín, khi phối hợp rbcL và matK thì dữ liệu thu được cho độ phân giải cao hơn so với từng gen riêng lẻ [75].
* Trình tự gen trnH-psbA
Gen trnH-psbA có kích thước trung bình khoảng 450 bp, nhưng thay đổi từ 296 đến 1120 bp, trnH-psbA được chứng minh là có khả năng xác định loài cao. Trình tự trnH-psbA đã được khuếch đại thành công ở nhiều thực vật hạt kín và hạt trần. Trong nhiều nghiên cứu gần đây đã đề xuất việc sử dụng trnH-psbA như chỉ thị ADN độc lập cho thực vật hay kết hợp với matK. CBOL thấy rằng khả năng phân biệt loài của trnH-psbA là cao nhất (69%) trong số 7 locus được thử nghiệm và do đó đề nghị nó như là chỉ thị ADN bổ sung [76].
1.4.4. Nghiên cứu về đa dạng di truyền loài Morinda officinalis How
1.4.4.1. Trên thế giới
Ding Ping và cộng sự đã chọn lọc ra 15 primer trong 100 primer ngẫu nhiên để nghiên cứu tính đa dạng di truyền của 5 quần thể Morinda officinalis (với 64 cá thể trồng tại Quảng Đông, Trung quốc) cho thấy có 112 mảnh đa hình cho thấy có sự biến đổi di truyền ở cấp độ loài [42].
Singh D.R. và cộng sự, đã nghiên cứu 52 primer RAPD và 60 primer ISSR để phân tích đa dạng di truyền của 3 loài trong chi Morinda là M. citrifolia, M. tinctoria và M. pubescens. Trong đó có 26 primer RAPD và 22 primer ISSR được khuếch đại và 11 primer đa hình [74]. Chỉ thị này cũng được Singh dùng để phân tích tính đa dạng của các mẫu của loài Morinda citrifolia được thu thập tại các vùng địa lý khác nhau [73].
Rahul R. Nair và cộng sự đã sử dụng kết hợp 2 gen matK và rcbL để phân biệt 2 loài Morinda reticulata Gamble và Morinda umbellata Linn (Rubiaceae) [67].
Đến thời điểm tháng 07/2017, Trên NCBI có trên 120 dữ liệu gen của loài Morinda officinalis [68]. Trong đó có khoảng 90 trình tự liên quan đến vùng ITS, 15 trình tự ở gen matK, 7 trình tự ở vùng gen rcbL, 5 trnH, 6 trnL và 1 trnT.
1.4.4.2. Tại Việt Nam
Hoàng Đăng Hiếu và cộng sự đã sử dụng primer ISSR để đánh giá mức độ đa dạng của 39 mẫu Ba kích thu được tại Quảng Ninh và kết luận rằng quần thể Ba kích tại Quảng Ninh đa dạng ở mức độ phân tử [7].
Năm 2018, Lê Hoàng Khẩn và cộng sự đã tiến hành nghiên cứu về sự đa dạng di truyền của cây Ba kích tại Quảng Nam. Nghiên cứu tiến hành trên 11 mẫu Ba kích thu hái tại Quảng Nam và Đà Nẵng. Kết quả nghiên cứu cho thấy có sự đa dạng di truyền dưới loài [9].
Định danh dược liệu bằng phương pháp chỉ thị ADN được một số nước thuộc khu vực Tây Thái Bình Dương thảo luận và thống nhất tiến tới xây dựng phương pháp và ngân hàng dữ liệu gen để áp dụng thông qua Diễn đàn FHH. Hiện tại, Dược điển Trung Quốc phiên bản năm 2015 đã có chuyên luận chung hướng dẫn định danh dược liệu bằng phương pháp chỉ thị ADN. Trong khi, tại Việt Nam đây là một lĩnh vực mới cần được nghiên cứu tiến tới bổ sung vào Dược điển.
1.6. Tính cấp thiết của luận án
Từ các thông tin tổng quan ở trên có thể thấy rằng, Ba kích là một dược liệu có nhiều tác dụng quý và có nhu cầu sử dụng lớn tại Việt Nam cần được kiểm soát chặt chẽ về chất lượng. Qua tham khảo tài liệu, monotropein và nystose là 2 “marker” của dược liệu rễ Ba kích do đây là những chất có hoạt tính sinh học và có hàm lượng lớn trong dược liệu này. Để kiểm soát tốt chất lượng dược liệu này, vấn đề đặt ra là phải nâng cấp chuyên luận dược liệu rễ Ba kích trong Dược điển Việt Nam V, trong đó
cần bổ sung chỉ tiêu định tính, định lượng 2 “marker” của dược liệu này (monotropein và nystose), đồng thời cần phân lập và thiết lập chất chuẩn monotropein và nystose nhằm mục đích cung cấp cho hệ thống kiểm nghiệm trên toàn quốc để giảm giá thành và thời gian cung ứng.
Như vậy, luận án "Nghiên cứu nâng cấp tiêu chuẩn dược liệu rễ Ba kích (Radix Morindae officinalis) của Việt Nam" được thực hiện với 3 mục tiêu chính như trên là cấp thiết.




![Nội Dung Thẩm Định Phương Pháp Định Lượng Nystose [52]](https://tailieuthamkhao.com/uploads/2024/04/20/nghien-cuu-nang-cap-tieu-chuan-duoc-lieu-re-ba-kich-radix-morindae-officinalis-7-120x90.jpg)
