Do có tới hai khả năng dẫn đến việc khuẩn lạc có màu trắng mà chúng tôi phải tiến hành PCR colony với mồi đặc hiệu vector M13 để chọn ra các dòng khuẩn lạc mang vector có đoạn gene cry1C được chèn vào.
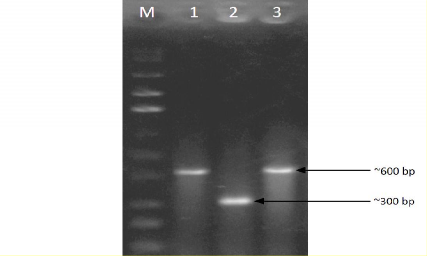
Hình 3.8 Điện di đồ sản phẩm PCR colony với mồi M13
1, 3: Sản phẩm PCR các dòng khuẩn lạc trắng TN 28.6 –p3, TN 6.12 –p7 2: Sản phẩm PCR dòng khuẩn lạc trắng TN 6.12 –p5
M: Thang DNA chuẩn
Điện di đồ sản phẩm PCR cho thấy DNA của các khuẩn lạc trắng TN 28.6
–p3, TN 6.12–p7 có kích thước khoảng 600 bp, phù hợp với tính toán lý thuyết (là kích thước của đoạn gene cry1C cộng với kích thước của sản phẩm PCR của vector bằng mồi M13). Trong khi đó ở khuẩn lạc trắng TN 6.12–p5 chỉ tổng hợp được đoạn gene có kích thước khoảng 300 bp (là kích thước của sản phẩm PCR của vector bằng mồi M13). Điều đó chứng tỏ các khuẩn lạc trắng TN 28.6–p3 và TN 6.12–p7 đã được biến nạp vector mang gene cry1C.
Có thể bạn quan tâm!
-
 Nghiên cứu đặc điểm sinh học của một số chủng Bacillus Thuringiensis sinh Protein tinh thể diệt côn trùng cánh vảy - 1
Nghiên cứu đặc điểm sinh học của một số chủng Bacillus Thuringiensis sinh Protein tinh thể diệt côn trùng cánh vảy - 1 -
 Nghiên cứu đặc điểm sinh học của một số chủng Bacillus Thuringiensis sinh Protein tinh thể diệt côn trùng cánh vảy - 2
Nghiên cứu đặc điểm sinh học của một số chủng Bacillus Thuringiensis sinh Protein tinh thể diệt côn trùng cánh vảy - 2 -
 Mô Hình Cấu Trúc Không Gian 3 Chiều Của Protein Độc Tố Cry1Ac
Mô Hình Cấu Trúc Không Gian 3 Chiều Của Protein Độc Tố Cry1Ac -
 Gene Cry1C Và Dưới Loài Bacillus Thuringiensis Subsp . Aizawai
Gene Cry1C Và Dưới Loài Bacillus Thuringiensis Subsp . Aizawai -
 Phân Loại Các Chủng Bt Bằng Phản Ứng Huyết Thanh
Phân Loại Các Chủng Bt Bằng Phản Ứng Huyết Thanh -
 Nghiên cứu đặc điểm sinh học của một số chủng Bacillus Thuringiensis sinh Protein tinh thể diệt côn trùng cánh vảy - 6
Nghiên cứu đặc điểm sinh học của một số chủng Bacillus Thuringiensis sinh Protein tinh thể diệt côn trùng cánh vảy - 6
Xem toàn bộ 63 trang tài liệu này.
Chúng tôi lấy các khuẩn lạc trắng đã sàng lọc bằng PCR colony và 1 khuẩn lạc xanh của từng chủng lần lượt cấy vào các lọ penicillin chứa 2ml môi trường LB bổ sung Amp (nồng độ 50 µg/ml) nuôi lắc ở 370C qua đêm để tách plasmid. DNA plasmid được kiểm tra bằng enzyme giới hạn. Do trong vùng MCS (multi cloning site) của vector pGEM–T Easy có chứa vị trí cắt của EcoRI nằm ở 2 đầu đoạn DNA insert nên chúng tôi cắt DNA plasmid bằng enzyme này
để kiểm tra sự có mặt của gene cry1C. Sản phẩm cắt được điện di trên gel agarose (Hình 3.9).
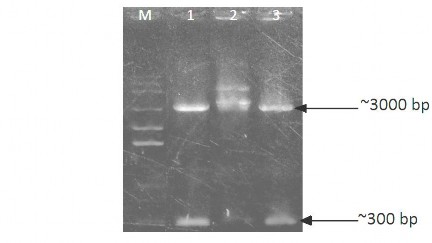
Hình 3.9. Điện di đồ phẩm cắt DNA plasmid tách chiết từ các dòng khuẩn lạc
Giếng 1,3. Sản phẩm cắt DNA plasmid dòng khuẩn lạc trắng của các plasmid tái tổ hợp pGEM-T Easy– cry1C – TN28.6- p3, pGEM-T Easy – cry1C – TN 6.12 – p7 Giếng 2. DNA plasmid dòng khuẩn lạc xanh (ĐC)
M:Thanh DNA chuẩn
Kết quả cho thấy DNA plasmid của các dòng khuẩn lạc trắng sau khi cắt bằng EcoRI tạo ra hai băng, băng lớn là vectơ tách dòng pGEM-T Easy có kích thước 3000 bp, băng nhỏ có kích thước gần 300 bp tương ứng với sản phẩm PCR của đoạn gene cry1C. Trong khi đó, plasmid của khuẩn lạc xanh sau phản ứng cắt chỉ cho 1 băng có kích thước khoảng 3000 bp (tương ứng với kích thước của vector tách dòng).
Để xác định chắc chắn sự có mặt của gene cry1C trong các khuẩn lạc trắng chúng tôi khuếch đại đoạn gene cry1C từ khuôn là plasmid thu được với cặp mồi đặc hiệu TYIC và TYIUNI (Hình 3.10).
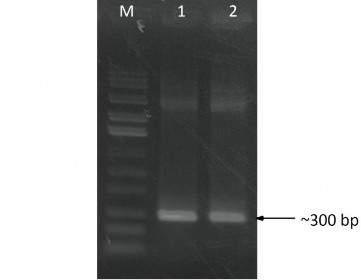
Hình 3.10. Điện di đồ sản phẩm PCR gene cry1C từ các DNA plasmid tái tổ hợp
pGEM-T Easy – cry1C – TN28.6 - p3, và pGEM-T Easy – cry1C – TN 6.12 – p7
Từ những kết quả thu được ở trên, chúng tôi có thể bước đầu kết luận là đã tách dòng thành công gene cry1C trong vector pGEM–T Easy. Tuy nhiên để có thể kết luận chính xác đoạn gene đã tách dòng có phải là đoạn gene cry1C hay không chúng tôi tiến hành đọc trình tự 2 đoạn gene này và so sánh với các trình tự gene trong Gene Bank.
3.2.3 Xác định trình tự đoạn gene cry1C
Plasmid tái tổ hợp pGEM-T Easy–cry1 –TN28.6 - p3 và pGEM-T Easy- cry1C–TN 6.12–p7 được chọn để đọc trình tự và được so sánh với trình tự có mã số AF362020 trên Gene Bank. Đoạn gene 288 bp của chủng TN6.12 có độ tương đồng 100% so với trình tự gene cry1C của chủng Bta C002 (AF362020) đã công bố trên Gene Bank. Trong khi đó , trình tự đoạn gen e cry1C của chủng TN 28.6 có độ tương đồng 99% so với chủng Bta C002 (AF362020) do bị mấ t 1 nucleotide ở vị trí 282.
Trình tự AF362020 là trình tự của gene cry1Ca mã hóa protein Cry1Ca. Như vậy có thể kết luận, trình tự gene cry1C của 2 chủng đã tách dòng thuộc phân nhóm gene cry1Ca.
Chương 4: KẾT LUẬN VÀ KIẾN NGHỊ
4.1. KẾT LUẬN
1. Từ 150 chủng Bacillus cereus chưa qua phân loại, đã xác định được 54 chủng B. thuringiensis sinh tinh thể, trong đó 26/54 chủng sinh 2 loại tinh thể dạng lưỡng tháp và cầu và được nhận dạng bằng type huyết thanh chuẩn. Trong đó có 10 chủng thuộc dưới loài B. thuringiensis subsp. aizawai chiếm tỷ lệ 38%
2. 7 chủng Bta có hoạt tính diệt sâu tơ và sâu xanh da láng, trong đó 4 chủng TN28.6, TN 1.12, TN6.12, và TN 36.3 có hoạt tính diệt sâu cao nhất.
3. Khuếch đại gene cry1C từ 7 chủng Bta đều cho sản phẩm có hoạt tính diệt sâu với cặp mồi đặc hiệu có kích thước 288 kb.
4. Đoạn gene cry1C từ 2 chủng TN 6.12 và TN 28.6 được tách dòng, đọc và so sánh với trình tự gene cry1C của chủng Bta có mã số AF362020 có độ tương đồng lần lượt là 100% và 99%. Các gene đã tách dòng thuộc phân nhóm gene cry1Ca
4.2. KIẾN NGHỊ
Gene cry1C được tách dòng từ 2 chủng TN6.12 và TN28.6 có hoạt tính diệt sâu cao, được xác định là đoạn gene cry1Ca mã hóa protein Cry1Ca. Trên cơ sở đó, thiết kế vector biểu hiện protein Cry1Ca nhằm nghiên cứu: sản xuất thuốc trừ sâu sinh học Bt diệt côn trùng bộ cánh vảy và làm nguyên liệu chuyển gene cry1C vào cây trồng tạo khả năng kháng lại côn trùng cánh vảy.
TÀI LIỆU THAM KHẢO
Tiếng Việt
1. Đái Duy Ban (2006), Công nghệ gen, Nhà xuất bản khoa học và kỹ thuật, trang 153.
2. Đặng Trọng Lương, Vũ Đức Quang, Nguyễn Hữu Đống, Trần Duy Quý (1999), Thiết kế lại cấu trúc gene Bt để chuyển vào cây hai lá mầm, Báo cáo Hội nghị sinh học toàn quốc, trang 1371-1376.
3. Hoàng Thị Lợi (2003), Giáo trình côn trùng học nông nghiệp đại cương tập 2, Nhà xuất bản Nông Nghiệp Hà Nội, trang 122 – 167.
4. Khuất Hữu Thanh (2004), Cơ sở di truyền phân tử và kỹ thuật gen, Nhà xuất bản khoa học và kỹ thuật, trang 109-147.
5. Ngô Đình Bính, Nguyễn Quỳnh Châu, Nguyễn Ánh Nguyệt (2002), Thu nhận huyết thanh miễn dịch cho phân loại Bacillus thuringiensis, Kỷ yếu Viện Công nghệ Sinh học 200-2001, trang 296-303.
6. Ngô Đình Bính, Nguyễn Quỳnh Châu, Nguyễn Văn Thưởng, Nguyễn Ánh Nguyệt, Trịnh Thế Cường, Jasser Mohamad Jamil, Ngô Đình Anh Trí, Nguyễn Hoài Trâm (2000), Nghiên cứu sự phân bố và đa dạng sinh học của Bacillus thuringiensis phân lập từ một số tỉnh ở Việt Nam. “Những vấn đề nghiên cứu cơ bản trong sinh học”. Báo cáo khoa học hội nghị sinh học quốc gia, trang 484- 488.
7. Ngô Đình Bính, Nguyễn Quỳnh Châu, Nguyễn Văn Thưởng, Trịnh Thị Ngọt, Nguyễn Ánh Nguyệt (2000), Sự phân bố của Bacillus thuringiensis trong các mẫu đất của Việt Nam, Tài nguyên sinh vật đất và sự phát triển bền vững của hệ sinh thái đất, trang 1-7.
8. Ngô Đình Bính (2005), Giáo trình thuốc trừ sâu sinh học.
9. Nguyễn Lân Dũng, Nguyễn Đình Quyến, Phạm Văn Ty (2000), Vi sinh vật học, Nxb Giáo dục.
10. Ngô Đình Bính, Lê Thị Minh Thành, Trịnh Thị Thu Hà, Phạm Kiều Thúy, Phạm Minh Hương, Nguyễn Thị Luy, Lê Thị Hồng Nhung, Đặng Văn Tiến (2010), “35 năm nghiên cứu và phát triển thuốc trừ sâu sinh học Bacillus thuringiensis tại Việt Nam”, Hội nghị khoa học kỷ niệm 35 năm Viện Khoa học và Công nghệ Việt Nam, tr. 288 – 300.
11. Lê Thị Minh Thành, Nguyễn Thị Thanh Hạnh, Nguyễn Xuân Cảnh, Nguyễn Ánh Nguyệt, Nguyễn Đình Tuấn, Phạm Kiều Thúy, Ngô Đình Bính (2005), Nghiên cứu sự phân bố và đa dạng gene của vi khuẩn Bacillus thuringiensis phân lập ở một số tỉnh thuộc vùng Bắc Bộ, Tạp chí Di truyền và ứng dụng, số 4, tr. 29 – 34.
12. Ngô Đình Bính, Nguyễn Quỳnh Châu, Nguyễn Ánh Nguyệt, Nguyễn Xuân Cảnh, Vi Thị Đoan Chính, Nguyễn Hoài Trâm, Nguyễn Văn Tuất (2003), “Tách dòng và biểu hiện gene mã hóa protein Cry1C diệt sâu khoang từ Bacillus thuringiensis subsp. aizawai” , Những vấn đề nghiên cứu cơ bản trong Khoa học Sự sống, Báo cáo khoa học hội nghị toàn quốc lần thứ hai, tr. 830 – 832.
13. Nguyễn Xuân Cảnh, Nguyễn Ánh Nguyệt, Nguyễn Thanh Hạnh, Nguyễn Quỳnh Châu, Ngô Đình Bính (2004), “ Nghiên cứu sự đa dạng sinh học của vi khuẩn Bacillus thuringiensis ở Việt Nam”, Báo cáo khoa học, nghiên cứu cơ bản trong Khoa học sự sống định hướng Nông lâm nghệp miền núi, Thái Nguyên 2004, NXB KHKT, tr. 59 – 62.
Tiếng Anh
14. Abad AR, Duck NB, Feng X, Flannagan RD, Kahn TW, Sims LE (2002)
Genes encoding novel protein with pesticidal activity against coleopterans.
15. Wu, Cao XL, Bai YY, and Aronson AI (1991), “Sequensing of an operon containing a novel δ-endotoxin gene from Bacillus thuringiensis’’. FEMS Microbi. Lett, page 31- 35.
16. Klier A (1985), “Biopesticide opportunities and challegene for anegement insect Parteun International Symposia”, page 16.
17. Tang W, Chen H, Xu C, Li, X., Lin, Y., Zhang X. (2007), “Development of insect – resistant transgenic indica rice with a synthetis Cry1C * gene” Mol. Breed, 18, pp. 1- 10
18. Asano S., Yamashita C., Iizuka T., Takeuchi K.,Yamanaka S., Cerf D., and Yamamoto S.(2003), “A strain of Bacillus thurigiensis subsp. galleriae contaning a novel cry8 gene highly toxic to Anomala cuprea”. Biological Control 28, page 191-196.
19. Lyon WaF. Confused and red flour beetles-HYG-2087-97, page 38.Ohio State University Extention Fact Sheet.
20. Crichmore N(2011), Bacillus thurigiensis Toxin Gene Nomenclature http://www.lifesci.sussex.ac.uk/home/Neil_Crickmore/Bt,27/07/2012.
21. Deacon J. "The microbial world: Bacillus thuringiensis",
www.helios.bto.ed.ac.uk/bto/microbes/Bacillus thuringiensis.htm 20/06/2012
22. Promega (2010), Technical Manual: pGEM®-T and pGEM®-T Easy Vector Systems, Instructions for use of products A1360, A1380, A3600, A3610, USA
23. Sambrook J and Russell D.W.(2001), Molecular Cloning - A laboratory Manual, Cold Spring Harbor Laboratory Press.
24. Schnepf E, Crickmore N., Van Rie N., Lereclus D., Baum F., Feitelson J., Zeigler D.R., and Dean D.H.,(1998) “Bacillus thuringiensis and its pesticidal crystal protein”. Microbiol Mol Biol Rev 62: page 775-806.
25. Burges H D (2001), "Bacillus thuringiensis in Pest control", Pesticide Outlook, pp. 90-98
26. Nester E.W, Thomashow L. S, Metz M. and Gordon M. (2002), 100 years of Bacillus thuringiensis: A Critical Scientific Assessment, American Academy of Microbiology,Washington, USA.
27. Theiry and E. Franchon (1997), “Identification, isolation, culture and preservation entomopathogenic bateria”, Biotechniques Manual of Technology in insect Pathology, Edited by Lawrence A. Lacey, Academic Press, pp. 55 – 57.