H | COOH | H | MeOH | |||
PPF | H | C6H5 | H | EG | ||
PMF | H | C6H5 | H | EG | ||
PAF | H | C6H5 | H | EG | ||
CPM | H | COOH | 4-MeO-C6H4 | EG | ||
CMM | H | COOH | 4-MeO-C6H4 | EG | ||
CIM | H | COOH | 4-MeO-C6H4 | EG | ||
PPM | H | C6H5 | 4-MeO-C6H4 | EG | ||
IPM | H | H | 4-MeO-C6H4 | EG | ||
BPM | Br | H | 4-MeO-C6H4 | EG | ||
BPC | Br | H | 4-Cl-C6H4 | EG | ||
Có thể bạn quan tâm!
-
 Tình Hình Nghiên Cứu Về Hoạt Tính Gây Độc Tế Bào Ung Thư Của Các Hợp Chất Có Khung Indole
Tình Hình Nghiên Cứu Về Hoạt Tính Gây Độc Tế Bào Ung Thư Của Các Hợp Chất Có Khung Indole -
 Phép Tam Giác Phân Đặt Ligand Vào Điểm Gắn Kết Của Protein
Phép Tam Giác Phân Đặt Ligand Vào Điểm Gắn Kết Của Protein -
 Các Dụng Cụ Và Thiết Bị Sử Dụng Trong Tổng Hợp
Các Dụng Cụ Và Thiết Bị Sử Dụng Trong Tổng Hợp -
 Docking Các Ligand Với Mục Tiêu Tác Động Và Đánh Giá Kết Quả
Docking Các Ligand Với Mục Tiêu Tác Động Và Đánh Giá Kết Quả -
 X Ch 3 ), 6,85 (2H, D, J = 8,5, H-3’ Và H-5’), 7,17 (2H, Dd, J = 3,0 Và J = 6,0, H-5
X Ch 3 ), 6,85 (2H, D, J = 8,5, H-3’ Và H-5’), 7,17 (2H, Dd, J = 3,0 Và J = 6,0, H-5 -
 (6)-Methyl-2-(2-Trifluoromethyl-Phenyl)-1H-Benzimidazole (2Tf)
(6)-Methyl-2-(2-Trifluoromethyl-Phenyl)-1H-Benzimidazole (2Tf)
Xem toàn bộ 186 trang tài liệu này.
(*) Ký hiệu tên các dẫn xuất indole gồm ba chữ cái biểu thị lần lượt cho 3 hợp phần indole, amine và aldehyde: indole C: indole-2-Carboxylic acid; P: 2-Phenylindole; I: Indole; B: 5-Bromoindole
amine P: Pyrrolidine; M: Morpholine; I: pIperidine; A: 1-methylpiperAzine aldehyde F: Formaldehyde; M: 4-Methoxybenzaldehyde; C: 4-Chlorobenzaldehyde
Các dẫn xuất được tổng hợp trong môi trường EG được thực hiện theo quy trình của Rajesh và cộng sự [118] (sơ đồ 2.3, bảng 2.4 và hình 2.3): cho vào bình cầu hai cổ 3 mmol dẫn xuất aldehyde và 6 mmol dẫn xuất amine bậc hai, hỗn hợp được khuấy thành dạng đồng nhất. Sau đó cho từ từ vào hỗn hợp 3 mmol dẫn xuất indole trong 5 mL ethylene glycol (EG) ở 60 °C. Hỗn hợp phản ứng được khuấy ở nhiệt độ phòng từ 24h - 36h và được theo dõi bằng sắc ký bản mỏng. Sau khi phản ứng kết
thúc, tủa rắn được thu bằng cách lọc dưới áp suất thấp. Chất rắn được rửa với nước và hexane, sau đó sấy khô thu được sản phẩm. Các sản phẩm CPF, CMF, CAF, CIF sử dụng dung môi phản ứng là methanol.
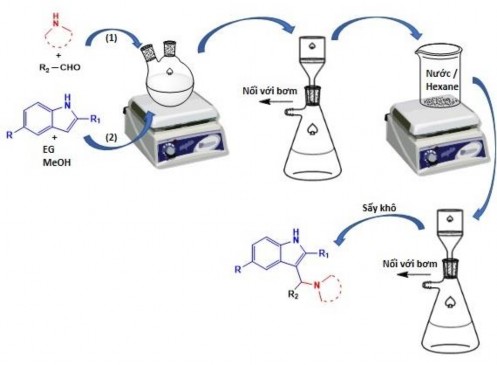
Hình 2.3. Các bước tổng hợp các dẫn xuất 3-aminoalkylated indole
Cơ chế phản ứng này được đề xuất như sau [118] (sơ đồ 2.4): EG có xu hướng tạo liên kết hydro liên phân tử và nội phân tử bằng hai nhóm hydroxyl liền kề [119, 120], giúp các phân tử tác chất trong phản ứng có cơ hội tiến lại gần nhau hơn nhờ tạo liên kết hydro liên phân tử. Nguyên tử hydro của EG có tính acid đã hoạt hóa nguyên tử oxygen giàu điện tử trong nhóm carbonyl của aldehyde, để hình thành giai đoạn trung gian (i). Một nguyên tử oxygen của EG có tính base hơn nguyên tử oxygen còn lại tấn công vào proton hoạt động của phân tử amine vòng, cùng lúc đó đôi điện tử tự do trên nguyên tử N của amine vòng tấn công vào nguyên tử carbon tích điện dương của nhóm carbonyl để tạo thành trạng thái trung gian (i và ii). Ở giai đoạn (ii), nguyên tử oxygen của EG kích hoạt proton hoạt động, tách loại một phân tử nước cùng với EG để tạo thành ion iminium ở giai đoạn (iii), ion iminium này tấn công vào vị trí C-3 giàu điện tử của phân tử indole, theo sau đó là sự chuyển dời nguyên tử hydro trong phân tử indole để tạo thành sản phẩm 3-alkylamino-1H-indole.2.3. Các phương pháp phân lập, xác định tính chất vật lý và cấu trúc các dẫn xuất benzimidazole và indole
![]()
Sơ đồ 2.4. Cơ chế phản ứng Mannich tạo các dẫn xuất 3-aminoalkylated indole
2.2.1. Sắc ký lớp mỏng (TLC)
Sắc ký lớp mỏng thực hiện trên bản silica gel tráng sẵn DC-Alufolien 60 F254 (Merck). Phát hiện chất bằng đèn tử ngoại Mineralight ® Lamp ở hai bước sóng 254 và 365 nm hoặc dùng thuốc thử là dung dịch H2SO4 10%/EtOH, sấy khô rồi hơ nóng trên bếp điện từ từ đến khi hiện màu.
2.2.2. Rửa nhiều lần bằng dung môi
Phương pháp này dựa trên nguyên tắc: chất phân cực sẽ tan trong dung môi phân cực và ngược lại. Tạp chất tan trong dung môi rửa mà không hòa tan chất cần phân lập, cần thiết phải lựa chọn dung môi phù hợp với độ phân cực và độ tan của chất trước khi tiến hành lọc rửa.
2.2.3. Kết tinh
Phương pháp kết tinh dựa trên sự khác nhau rõ rệt về độ tan của các chất trong một dung môi (hay hỗn hợp các dung môi) ở các nhiệt độ khác nhau, hoặc có sự khác nhau về độ tan giữa chất chính và tạp chất ở cùng một nhiệt độ. Dung môi hoặc hệ dung môi được lựa chọn để phù hợp kết tinh với từng chất.
2.2.4. Sắc ký cột (CC)
Sắc ký cột được tiến hành với chất hấp phụ là silica gel pha thường 230-240 mesh (Merck) với hệ dung môi giải ly khác nhau phù hợp với từng chất.
2.2.4. Sắc ký lỏng hiệu năng cao (HPLC)
Dữ liệu sắc ký lỏng cao hiệu năng cao được bổ sung cho một hợp chất có hoạt tính sinh học tốt như 5MM, 53H, BPM, PPM, 3TF: sử dụng hệ máy HPLC Agilent 1200 (Agilent Technologies, CA, USA), đầu dò DAD ở bước sóng 254 nm, dữ liệu được ghi nhận bởi phần mềm Agilent Chemstation 4.0. Sử dụng cột C18 pha đảo Zorbax SB-C18 (Agilent Technologies, CA, USA) với thông số cột 4.6 mm×250 mm, kích thước hạt 5 μm. Thực hiện phân tích ở 26 °C, tốc độ dòng 0,8 mL/phút trong suốt quá trình chạy. Dung môi rửa giải A: nước cất chứa 0,1% formic acid và B: 100% acetonitrile. Điều kiện chạy như sau:
Dung môi A (%) | Dung môi B (%) | |
0 | 70 | 30 |
20 | 30 | 70 |
21 | 0 | 100 |
27 | 0 | 100 |
2.2.5. Đo điểm nóng chảy
Điểm nóng chảy được đo trên máy Electrothermal 9100 (UK), dùng mao quản, không hiệu chỉnh tại Viện Công nghệ Hóa học – Viện Hàn lâm Khoa học và Công nghệ Việt Nam.
2.2.6. Phương pháp phổ hồng ngoại (FTIR)
Phổ hồng ngoại được đo trên máy FTIR Bruker Equinox 55, với kỹ thuật viên nén KBr trong vùng 4000- 400 cm-1, tại Viện Công nghệ Hóa học – Viện Hàn lâm Khoa học và Công nghệ Việt Nam.
2.2.7. Phương pháp khối phổ phân giải cao (HRMS)
Khối phổ phân giải cao (dùng kĩ thuật ESI: electrospray ionization) được ghi trên hệ thống máy X500R QTOF (Sciex) tại trường Đại học Khoa học Tự nhiên – Đại học Quốc gia Tp.HCM.
2.2.8. Phương pháp phổ cộng hưởng từ hạt nhân (NMR)
Phổ cộng hưởng từ hạt nhân (NMR) được đo trên máy Bruker Advance AV500 MHz và Bruker DRX 500, tần số 500 MHz với phổ 1H-NMR và 125 MHz đối với phổ 13C-NMR, tại Viện Hóa Học – Viện Hàn lâm Khoa học và Công nghệ Việt Nam.
2.3. Phương pháp xác định hoạt tính gây độc tế bào in vitro của các dẫn xuất benzimidazole và indole
Ba dòng tế bào ung thư trên người sử dụng trong nghiên cứu: A549 (ung thư phổi), MDA-MB-231 (ung thư vú), PC3 (ung thư tuyến tiền liệt) được cung cấp bởi giáo sư Jeong-Hyung Lee – Khoa Khoa học Tự nhiên – Đại học Quốc gia Kangwon
– Hàn Quốc. Thử nghiệm ức chế tế bào ung thư được thực hiện tại Trung tâm tiên tiến về hóa sinh hữu cơ – Viện Hóa Sinh Biển – Viện Hàn lâm Khoa học và Công nghệ Việt Nam. Thử nghiệm gây độc dòng tế bào HEK 293 (tế bào thận gốc phôi ở người) – có nguồn gốc từ Ngân hàng Tế bào Hoa Kỳ ATCC – được thực hiện tại Viện Công nghệ Sinh học - Viện Hàn lâm Khoa học và Công nghệ Việt Nam.
2.3.1. Nguyên tắc xác định
Các dẫn xuất tổng hợp benzimidazole và indole được đánh giá khả năng gây độc tế bào bằng phương pháp MTT (3-(4,5-dimethythiazol-2-yl)-2,5-diphenyl tetrazolium bromide) trên các dòng tế bào ung thư ở người: ung thư phổi A549, ung thư vú MDA-MB-231, ung thư tuyến tiền liệt PC3, sử dụng chứng dương là camptothecin. Nguyên lý của phép thử là vòng tetrazolium bám chặt vào ty thể của tế bào hoạt động, dưới tác dụng của enzyme succinate dehydrogenase (SDH) của ty thể chỉ có trong tế bào sống, MTT (màu vàng) biến đổi thành các tinh thể formazan (màu tím) (sơ đồ 2.5). Số lượng tế bào sống tỉ lệ thuận với nồng độ formazan, thể hiện qua giá trị mật độ quang OD của dung dịch đo ở 570 nm [121]. Độ hấp thụ được đo trên hệ máy ELISA Bio-Rad (Mỹ) tại Trung tâm tiên tiến về hóa sinh hữu cơ – Viện Hóa Sinh Biển – Viện Hàn lâm Khoa học và Công nghệ Việt Nam.
![]()
Muối tetrazolium MTT (màu vàng) Formazan (màu tím)
Sơ đồ 2.5. Sự chuyển hoá của muối MTT dưới tác động của ty thể
2.3.2. Cách tiến hành
Các tế bào ung thư được nuôi cấy in vitro theo phương pháp của Tim Mosmann và cộng sự [122]. Tế bào được nuôi cấy 48h trong môi trường RPMI 1640 hoặc DMEM ở 37 oC, 5% CO2 với 10% FBS, penicillin (100 unit/mL) và streptomycin (100µg/mL). Sau đó chúng được nuôi cấy trong đĩa 96 giếng với thể tích là 200 µl, mật độ 2-5x105 tế bào/giếng (tuỳ từng loại tế bào). Sau 24h, chúng được thử với hợp chất pha sẵn ở các nồng độ khác nhau trong DMSO. Sau 72h, cho phản ứng với 0,5 mg/µL MTT, ủ 4h ở 37oC và 5% CO2. Sau đó hút bỏ hết môi trường trên bề mặt, kết tủa formazan được hòa tan trong isopropanol. Camptothecin (CPT) được sử dụng làm chứng dương.
2.3.3. Đánh giá kết quả
OD (mẫu thử) – OD ( ngày 0)
𝐶𝑆% = [
OD (DMSO) – OD ( ngày 0)
× 100] ± 𝑆𝐷
Trong đó: CS% (Cell Survival%): tỉ lệ sống sót của tế bào; OD: mật độ quang; SD (Standard Deviation): độ lệch tiêu chuẩn, được tính bằng công thức bên dưới.
√(∑ 𝑥𝑖 − 𝑥̅)2
𝑆𝐷 =
𝑛 − 1
Trong đó: xi: giá trị OD tại giếng i; 𝑥̅: giá trị OD trung bình; n: số giếng thử lặp lại Các mẫu có biểu hiện hoạt tính (CS ≤ 50% ± SD) sẽ được chọn ra cho bước
thử nghiệm tiếp theo.
2.4. Xây dựng mô hình docking phân tử
Các phần mềm và thiết bị sử dụng trong quá trình docking được liệt kê chi tiết ở bảng 2.5.
Bảng 2.5. Các phần mềm và thiết bị sử dụng trong quá trình docking
Mục đích sử dụng | Thương mại | |
ChemDraw 19.1 [123] | Xây dựng cấu trúc 2D | X |
MOE 2015.10 [124] | Chuyển đổi cấu trúc 2D sang 3D, và tối ưu hóa cấu trúc 3D của các hợp chất (tối thiểu hóa năng lượng). | X |
Chuẩn bị và tối ưu hóa cấu trúc protein cho quá trình docking. | ||
Sybyl-X 1.1 [125] | Mô phỏng động lực học phân tử của các hợp chất để chọn ra được cấu trúc có năng lượng tối thiểu toàn phần | X |
BiosolveIT LeadIT 2.1.8 [126] | Docking phân tử | X |
Discovery Studio Visualizer 4.0 [127] | Phân tích tương tác và trình bày cấu trúc sau khi docking | |
Thiết bị | ||
Tất cả các quá trình xây dựng mô hình docking được thực hiện trên hệ thống máy vi tính có cấu hình Intel(R) Core(TM) i3-9100F CPU @ 3,60GHz, 8GB RAM, Visual Graphic Card: NVIDIA GeForce GTX 1650 4GB và trên hệ điều hành 64 bit Windows 10 (Microsoft, Redmond, WA, USA) | ||
Quy trình xây dựng mô hình docking gồm các bước sau:
Bước 1
• Chuẩn bị cơ sở dữ liệu
Bước 2
• Docking lặp lại (Re-docking)
Bước 3
• Docking các ligand với mục tiêu tác động
Bước 4
• Đánh giá kết quả từ mô hình
2.4.1. Chuẩn bị cơ sở dữ liệu
Các cấu tử được vẽ công thức cấu tạo bởi chương trình ChemDraw Ultra 19.1. Tiếp tục được tối ưu hoá năng lượng bằng chương trình MOE 2015.10. Tối ưu hóa năng lượng lần 2 được thực hiện bởi chương trình Sybyl-X 1.1 nhằm đưa ra được cấu dạng có năng lượng tối thiểu trong toàn bộ các cấu dạng được tạo ra. Vì các cấu dạng ban đầu được vẽ bằng phần mềm ChemDraw nên độ dài các liên kết, vị trí các nguyên
tử, góc liên kết có thể không phù hợp với thực tế. Do đó việc tối ưu hóa năng lượng sẽ quay lại các góc liên kết, đặt lại vị trí các nguyên tử, điều chỉnh độ dài các liên kết nhằm tạo ra cấu dạng có năng lượng tối thiểu. Cấu dạng có năng lượng tối thiểu thường bền hơn các cấu dạng có năng lượng cao hơn. Việc chọn lựa cấu dạng có năng lượng tối thiểu nhằm nâng cao khả năng tương thích giữa kết quả docking và khả năng thể hiện hoạt tính thực tế của cấu tử.
2.5.1.1. Chuẩn bị cấu trúc protein
Cấu trúc protein được chuẩn bị trong MOE 2015.10 bằng công cụ Sequence Editor và QuickPrep. Phức hợp protein được sử dụng trong nghiên cứu docking đối với các dẫn xuất benzimidazole là phức hợp enzyme topoisomerase I (TopI) DNA của người với CPT và cộng hóa trị với một chuỗi DNA xoắn kép, được tải về từ ngân hàng protein (RCSB Protein Data Bank), ký hiệu là 1T8I. Nước kết tinh (HOH) và ion (Na+, Cl-…) trong dung môi cũng sẽ được xoá bỏ. Hình 2.4 cho thấy ligand đồng kết tinh tương tác tốt với cấu trúc khoang gắn kết được tạo ra để tiến hành docking.
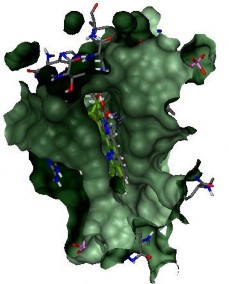
Hình 2.4. Cấu trúc khoang gắn kết của phức hợp TopI và chuỗi DNA xoắn kép với ligand CPT
2.5.1.2. Chuẩn bị cấu trúc ligand
Cấu trúc 2D của các ligand được chuẩn bị bằng phần mềm Chemdraw 19.1 và lưu file có định dạng *.mol2 để tiến hành bước xử lý tối ưu thiểu hóa năng lượng trong Sybyl-X 2.0. Các bước chuẩn bị trong Sybyl-X 2.0 bao gồm tối thiểu hóa năng lượng lần 1, chạy động lực học, sau đó tối thiểu hóa năng lượng lần 2. Các thông số tối thiểu hóa năng lượng cho cả lần 1 và 2 được thiết lập như sau: