27
Design) [25, 76, 77]. Hiện nay, phương pháp docking bắt đầu được sử dụng phổ biến vì tính đơn giản, dễ sử dụng và ít tốn thời gian trong các phương pháp sàng lọc ảo. Phương pháp này được dùng để đánh giá và dự đoán khả năng gắn kết hoặc ái lực liên kết giữa các phân tử ligand và mục tiêu tác động (protein, enzyme, DNA) thông qua điểm số docking cũng như các vị trí gắn kết (binding site, active site).
1.4.1.1. Phân loại
Docking tìm các tương tác giữa các ligand và mục tiêu tác động theo ba hướng sau [78]:
- Xem ligand và mục tiêu tác động đều là những phân tử cứng (docking cứng).
- Xem ligand là những phân tử linh động và mục tiêu tác động là những phân tử cứng (docking bán linh động).
- Xem cả ligand và mục tiêu tác động đều là những phân tử linh động (docking linh động hoàn toàn), nhưng tính linh động chỉ giới hạn ở những chuỗi bên đặc trưng nào đó của điểm gắn kết.
1.4.1.2. Phần mềm FlexX trong docking
Trong hai thập kỷ gần đây có khoảng 60 công cụ và phần mềm khác nhau được phát triển cho quá trình docking phân tử [79], trong đó phổ biến nhất là các phần mềm như: DOCK [80], AutoDock [81], FlexX [82], Surflex [83], GOLD [84], ICM [85],
Glide [56], Cdocker, LigandFit [80], MCDock, FRED [86], MOE-Dock [87], LeDock [64], AutoDock Vina [88], rDock [89], UCSF Dock [90]… Tận dụng nguồn tài nguyên sẵn có, trong nghiên cứu này chương trình FlexX trong bộ phần mềm LeadIT 2.1.8 được sử dụng trong quá trình docking.
Có thể bạn quan tâm!
-
 Một Số Thuốc Điều Trị Ung Thư Chứa Khung Benzimidazole
Một Số Thuốc Điều Trị Ung Thư Chứa Khung Benzimidazole -
 Cấu Trúc Của Các Dẫn Xuất Benzimidazole- 1,3,4-Oxadiazole
Cấu Trúc Của Các Dẫn Xuất Benzimidazole- 1,3,4-Oxadiazole -
 Tình Hình Nghiên Cứu Về Hoạt Tính Gây Độc Tế Bào Ung Thư Của Các Hợp Chất Có Khung Indole
Tình Hình Nghiên Cứu Về Hoạt Tính Gây Độc Tế Bào Ung Thư Của Các Hợp Chất Có Khung Indole -
 Các Dụng Cụ Và Thiết Bị Sử Dụng Trong Tổng Hợp
Các Dụng Cụ Và Thiết Bị Sử Dụng Trong Tổng Hợp -
 Các Bước Tổng Hợp Các Dẫn Xuất 3-Aminoalkylated Indole
Các Bước Tổng Hợp Các Dẫn Xuất 3-Aminoalkylated Indole -
 Docking Các Ligand Với Mục Tiêu Tác Động Và Đánh Giá Kết Quả
Docking Các Ligand Với Mục Tiêu Tác Động Và Đánh Giá Kết Quả
Xem toàn bộ 186 trang tài liệu này.
FlexX là chương trình docking được Rarey và cộng sự giới thiệu năm 1996 [82], dự đoán tương tác giữa phức hợp protein và ligand thông qua cấu trúc hình học và ái lực gắn kết của phức hợp này trong đó protein được giữ cứng. Thuật toán docking trong FlexX hoạt động không cần sự can thiệp thủ công. Đặc biệt FlexX rất hữu dụng trong trường hợp đã có cấu trúc 3D của protein và vị trí hoạt động cụ thể của protein, có tập hợp phân tử các chất, FlexX lúc này sẽ phân tích việc gắn kết của protein với mỗi chất kể trên.
Trong FlexX, cấu trúc của mục tiêu (protein) được giữ cứng, còn cấu dạng của ligand được thiết kế linh động bằng cách tạo ra tập hợp nhiều cấu dạng khác nhau của ligand. Các tương tác ligand – mục tiêu được tính toán bao gồm liên kết hydro, liên
kết kim loại và tương tác kỵ nước. Thuật toán docking của FlexX dựa trên cơ sở chiến lược xây dựng gia tăng (hình 1.5), gồm 3 bước như sau [82, 91, 92]:
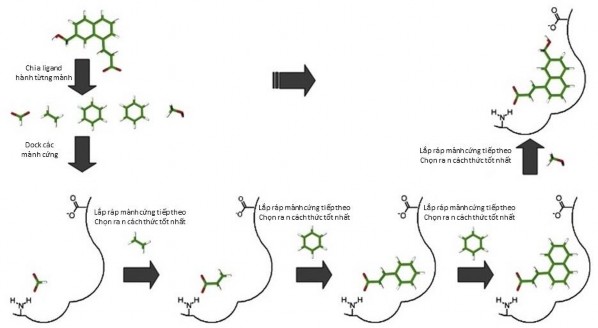
Hình 1.5. Thuật toán docking xây dựng gia tăng
Lựa chọn mảnh cơ sở: mảnh cơ sở được tách ra từ ligand sao cho số lượng nhóm có khả năng tạo tương tác là lớn nhất và số lượng cấu dạng của mảnh cơ sở trong vùng gắn kết là nhỏ nhất. Sau khi lựa chọn được mảnh cơ sở, phần còn lại của ligand được tự động chia thành nhiều mảnh.
Định vị mảnh cơ sở: việc định vị mảnh cơ sở dựa trên thuật toán tam giác phân. Bề mặt vùng tương tác của mục tiêu được chuyển thành một tập hợp giới hạn các điểm tương tác, còn mảnh cơ sở được phân tích thành các trung tâm tương tác. Mảnh cơ sở được định vị vào vùng hoạt động bằng cách ánh xạ từng cặp 3 trung tâm tương tác của mảnh cơ sở lên 3 điểm tương tác của mục tiêu. Các vị trí của mảnh cơ sở được tập hợp lại và sàng lọc để tập hợp lại theo nhóm các vị trí có tương tác gần giống nhau với mục tiêu hay cùng một cấu dạng nhưng tạo được tương tác với nhiều bộ ba điểm tương tác khác nhau. Cuối cùng, các vị trí tốt nhất của mảnh cơ sở được lựa chọn từ các nhóm trên.
Xây dựng cấu dạng gắn kết của ligand: từ tập hợp vị trí của các mảnh cơ sở, các mảnh còn lại của ligand được lắp ráp theo thứ tự vào mảnh cơ sở để tìm ra những vị trí có năng lượng liên kết thấp nhất. Quá trình này được thực hiện bằng thuật toán xây dựng gia tăng.
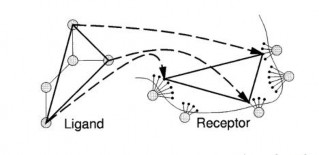
Hình 1.6. Phép tam giác phân đặt ligand vào điểm gắn kết của protein
1.4.2. Tổng quan về phức hợp camptothecin-topoisomerase I-DNA
1.4.2.1. Giới thiệu chung
Topoiosmerase-DNA là nhóm enzyme phổ biến nhất trong tất cả các vật thể sống từ virus tới con người, chịu trách nhiệm đóng và tháo xoắn phân tử DNA, tạo điều kiện cho các enzyme khác cùng phối hợp trong quá trình sao chép, nhân đôi, phiên mã và các quá trình khác trong hạt nhân [93]. Chúng thực hiện chức năng này bằng cách cắt liên kết phosphodiester – xương sống của DNA – gỡ bỏ cuộn xoắn của DNA, sau đó hình thành hợp chất trung gian cộng hóa trị phosphotyrosine với DNA [94]. Các enzyme topoisomerase được chia thành hai nhóm lớn dựa trên chức năng chính của từng loại: topoisomerase I (TopI) tháo xoắn một sợi của chuỗi xoắn kép DNA, topoisomerase II (TopII) tháo xoắn hai sợi của chuỗi xoắn kép DNA (hình 1.1). Các thuốc chống ung thư nhắm mục tiêu là enzyme topoisomerase có tác dụng gây độc enzyme topoisomearase, dẫn đến quá trình tháo xoắn bị dừng lại và cuối cùng là đứt gãy chuỗi xoắn kép DNA [95].
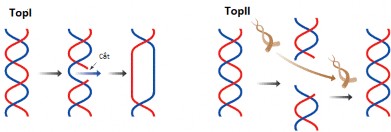
(Nguồn: Internet)
Hình 1.7. Mô tả enzyme topoisomerase I (TopI) và topoisomerase II (TopII) tháo xoắn chuỗi DNA
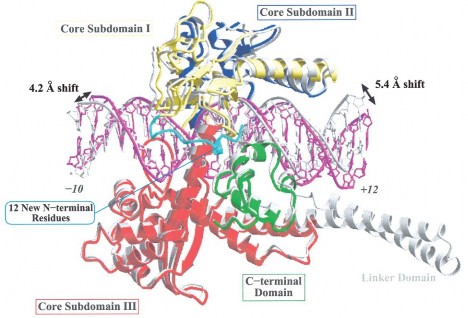
(Nguồn: Redinbo [96]) Miền đầu tận cùng NH2 (màu xanh lơ), miền lõi gồm 3 miền phụ core subdomain I-III (màu vàng, xanh dương và đỏ), miền liên kết (màu trắng), miền đầu tận cùng COOH (màu xanh lá), chuỗi xoắn kép DNA
Hình 1.8. Cấu trúc của phức hợp enzyme TopI-DNA người (TopI-DNA)
Cấu trúc của phức hợp enzyme TopI-DNA ở người bao gồm chuỗi xoắn kép DNA và enzyme TopI (hình 1.8). Enzyme TopI có trọng lượng phân tử khoảng 93 kDa, bao gồm 4 miền chính: miền đầu tận cùng NH2 24 kDa (N-terminal domain), miền lõi 56 kDa (core domain – gồm 3 miền lõi phụ core subdomain I-III), miền liên kết 7 kDa (linkder domain) và miền đầu tận cùng COOH 6 kDa (C-terminal domain) chứa amino acid xúc tác Tyr-723 [97]. Trong đó miền đầu tận cùng NH2 đóng vai trò không thể thiếu trong hoạt động xúc tác của phức hợp enzyme [97]. Trong phức hợp này, enzyme TopI bao bọc xung quanh chuỗi xoắn kép DNA do đó giữa enzyme TopI và DNA tương tác với nhau bằng nhiều liên kết, đa phần là liên kết phosphate [98, 99]. Tham gia vào quá trình hoạt động xúc tác của phức hợp là các amino acid quan trọng Lys532, Tyr723, Arg488, Arg590 và His632 nằm ở vùng hoạt động (active site) được giới hạn bởi miền đầu tận cùng NH2, COOH và chuỗi xoắn kép DNA. Lys523 gắn với nguyên tử oxy của base thymine10 trên chuỗi xoắn (ở phần bị cắt đứt bởi enzyme TopI), Tyr723 tấn công vào nguyên tử phospho của nhóm phosphate nằm trên chuỗi xoắn, trong khi đó các amino acid Arg488, Arg590 và His632 có tác dụng làm ổn định trạng thái chuyển tiếp nhóm phosphate của chuỗi [96, 99].
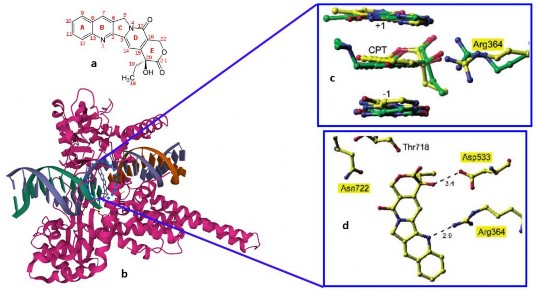
(Nguồn: Wall và Staker [100, 101]) Hình 1.9. (a) Cấu trúc của CPT, (b) Cấu trúc ba chiều của phức hợp bậc ba CPT-
TopI-DNA, (c) CPT xen vào chuỗi xoắn DNA và (d) các amino acid ở vùng hoạt động của CPT trong phức hợp
Camptothecin (CPT) được biết đến là hợp chất tự nhiên có khả năng ức chế khối u cao, theo cơ chế ức chế phức hợp enzyme TopI-DNA [102, 103]. CPT gắn kết vào phức hợp TopI-DNA tạo thành phức hợp bậc 3 bền CPT-TopI-DNA (ternary complex) [101], TopI-DNA bị kẹt lại không thể tiếp tục thực hiện chức năng tháo xoắn chuỗi DNA [104, 105] (hình 1.9). Cấu trúc tinh thể của phức hợp bậc 3 này cho thấy CPT xen vào chuỗi xoắn của phần DNA phân tách, nằm cách 3.8 đến 4.0 Å và tương tác với các amino acid Lys532, Tyr723 và thymine10 ở vùng hoạt động của TopI-DNA. Nguyên tử oxy ở C17 tương tác và cách 4.0 Å với Asn722. Trong khi đó, nhóm hydroxyl ở C-20 tạo liên kết hydro với Asp533 – đặc trưng cho độ nhạy của enzyme với CPT [105]. Tương tác gần nhất của CPT với enzyme là với Arg364, cách
2.9 Å.
1.4.2.2. Tình hình nghiên cứu mô hình docking của các hợp chất có khung benzimidazole và indole trên đối tượng tác động phức hợp TopI-DNA
a) Khung benzimidazole
Các nghiên cứu cho thấy khung cấu trúc benzimidazole đã được nhìn nhận như là những tác nhân ức chế tế bào ung thư và đặc biệt có đích tác động là phức hợp enzyme topoisomerase I-DNA [106-108]. Cụ thể đã có các nghiên cứu về khả năng ức chế TopI-DNA trên các dẫn xuất 2-phenylbenzimidazole 2 và 9 (J. La Voie et al,
1996 [109]), dẫn xuất I-III (Z. Topcu et al, 2007 [110]), các bis-benzimidazole
Hoechst 33458 và Hoechst 33342 (R. Kakkar et al, 2019 [111]) (hình 1.10).
![]()
![]()
Hình 1.10. Các tác nhân ức chế phức hợp Topoisomerase I-DNA có chứa khung benzimidazole
Song song đó là các nghiên cứu sử dụng mô hình docking để giải thích và hiểu rõ hơn cơ chế gắn kết của các dẫn xuất benzimidazole vào phức hợp TopI-DNA ở mức độ phân tử. Để chứng minh hợp chất 5n có khả năng ức chế TopI-DNA tương tự như camptothecin và Hoechst 33342, Cevik (năm 2020) đã nghiên cứu tương tác của 5n với phức hợp TopI-DNA bằng mô hình docking (PDB ID: 1T8I), sử dụng phần mềm Glide 7.1 (hình 1.11).
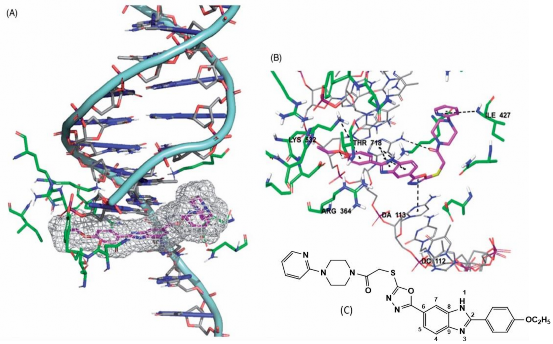
(Nguồn: Cevik, 2020 [43]) Hình 1.11. Kết quả docking của 5n: (A) 5n nằm trong khoang hoạt động của phức hợp TopI-DNA, (B) Các tương tác chính của 5n với khoang hoạt động của phức
hợp TopI-DNA, (C) Cấu trúc 5n
Kết quả cho thấy 5n tương tác với DNA và ngăn không cho TopI gắn kết vào DNA. Cụ thể 5n có tương tác với các amino acid chính Glu356, Arg364, Lys425, Arg488, Lys532, Asp533, Thr718 trong khoang hoạt động của phức hợp TopI-DNA bằng liên kết hydro và lực van der Waals, ngoài ra còn tương tác với các base da113, dc112 và dt10 của chuỗi DNA [43].
b) Khung indole
![]()
![]()
Các nghiên cứu cho thấy khung cấu trúc indole đã được nhìn nhận như một trong những tác nhân ức chế topoisomerase I-DNA, cụ thể như hợp chất evodiamine chứa khung nền indole trong phân tử đã được chứng minh ức chế chọn lọc phức hợp TopI-DNA (A. L.-F Chan et al, 2009 [112]), hợp chất 18a (R. Chaniyara et al, 2013 [113]), dẫn xuất 4n (C. Zhang et al, 2016 [114]), dẫn xuất 6n (B. Shu et al, 2020 [115]) (hình 1.12).
Hình 1.12. Các tác nhân ức chế phức hợp Topoisomerase I-DNA có chứa khung indole
Song song đó là các nghiên cứu trên mô hình docking như nghiên cứu của Wannian Zhang (2010) trên các dẫn xuất của evodiamine cho thấy hợp chất 29u có khả năng ức chế tốt trên 3 dòng tế bào thử nghiệm A549, MDA-MB-231 và HCT116 (IC50 lần lượt là 0,86; 0,049 và 2,6 µM), tương đương và có khả năng ức chế tốt hơn CPT (IC50 lần lượt là 0,057; 3,1 và 0,71 µM). Hợp chất 29u được tiếp tục nghiên cứu bằng mô hình docking để hiểu rõ khả năng gắn kết ở mức độ phân tử với phức hợp
TopI-DNA (PDB ID: 1T8I), kết quả cho thấy 29u nằm trong khoang hoạt động của TopI-DNA và có cách thức gắn kết giống với CPT khi hình thành tương tác với chuỗi DNA, ngoài ra nó còn hình thành tương tác với phức hợp bằng liên kết hydro với amino acid Arg364 [116] (hình 1.13).
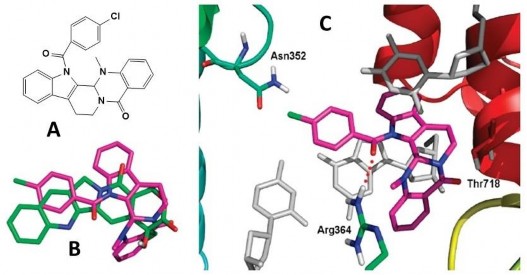
(Nguồn: Wannian Zhang, 2010 [116]) Hình 1.13. (A) Cấu trúc của dẫn xuất evodiamine 29u (B) Cấu hình docking của 29u và CPT được chồng lên (C) 29u gắn kết vào khoang hoạt động của TopI-DNA
Dựa vào các phân tích trên, nhóm nghiên cứu đã đề xuất một trong những cơ chế ức chế ở mức độ phân tử đối với tế bào ung thư của các dẫn xuất benzimidazole và indole có hoạt tính tốt nhất: đích tác động phức hợp topI-DNA (PDB: 1T8I) và so sánh với đối chứng dương camptothecin để làm rõ cách thức gắn kết của các dẫn xuất này vào phức hợp TopI-DNA.