chu trình Applied Biosystems Big Dye Terminator (Ứng dụng Biosytems, Foster City, CA, USA) tại Viện Công nghệ Sinh học, Hà Nội, Việt Nam.
2.3.5 Chuyển gen tạo cây đậu tương mang cấu trúc CRISPR/Cas9
Phương pháp chuyển gen vào cây đậu tương thông qua vi khuẩn A. tumefaciens được thực hiện dựa trên quy trình chuẩn tại phòng Công nghệ tế bào thực vật Viện Công nghệ sinh học (2020) [90] với các môi trường sử dụng trong thí nghiệm (Bảng 2.2) và bước thực hiện cơ bản như sau:
Bảng 2.2 Thành phần các môi trường sử dụng trong chuyển gen cây đậu tương
Thành phần | pH | |
GCM (gieo hạt) | Muối đa lượng MS (10X) 100ml + muối vi lượng MS (100X) 10ml + Fe-NaEDTA (100X) 10ml + sucrose 20g/l + Agar 7 g/l | 5,6 |
YEP lỏng (nuôi huyền phù khuẩn) | L-triptone or peptone 10g/l + Yeast Extract 10g/l + NaCl 5g/l (Nếu là môi trường đặc bổ sung Bacto agar 12 g/l) | 7,0 |
- CCM lỏng | Muối đa lượng B5 (10X) 10ml + Muối vi lượng B5 (100X) 1ml | |
(hòa khuẩn | + Fe-NaEDTA (100X) 1ml + MES (20Mm) 3,9 g/l + sucrose | |
dùng biến | 30g/l + Acetosyringone 0,04g/l + Viatmin B5 (100X) 10ml + | |
nạp) | GA3 (1mg/ml) 0,25ml + BAP (1,7mg/ml) 1ml + L-cystein | 5,4 |
- CCM đặc | 0,4g/l + Dithiothreitol (DTT) 0,154g/l + Na2SiO3 0,158g/l | |
(đồng nuôi | (Nếu là môi trường đặc bổ sung Agar 5g/l) | |
cấy) | ||
- SIM lỏng | Muối đa lượng B5 (10X) 100ml + muối vi lượng B5 (100X) | |
(diệt khuẩn) | 10ml + Fe-NaEDTA (100X) 10ml + MES (3 Mm) 0,6g/l + | 5,7 |
sucrose 30g/l + Viatmin B5 (100X) 10ml + BAP (1,7mg/ml) | ||
- SIM đặc | 1ml + Timetin 50mg/l + Glufosinate 5mg/l | |
(tạo đa chồi) | (Nếu là môi trường đặc bổ sung Phytagel 3g/l) | |
SEM | Muối đa lượng B5 (10X) 100ml + muối vi lượng B5 (100X) | |
(môi trường | 10ml + Fe-NaEDTA (100X) 10ml + MES (3 Mm) 0,6g/l + | |
kéo dài | sucrose 30g/l + Viatmin B5 (100X) 10ml + GA3 (1mg/ml) | |
chồi) | 0,5ml + Timetin 50mg/l + Cefotaxime 100 mg/l + IAA | |
(1mg/ml) 100 microlit + Zeatin-R (1mg/ml) 1ml + Glufosinate | ||
2 mg/l | ||
RM (tạo rễ) | Muối đa lượng MS (10X) 100ml + muối vi lượng MS (100X) 10ml + Fe-NaEDTA (100X) 10ml + MES (3 Mm) 0,6g/l + sucrose 20g/l + Viatmin B5 (100X) 10ml + BAP (1,7 mg/ml) 1ml + Timetin 50 mg/l + Asp/Glu (stock 5 mg/ml) 10ml + Cefotaxime 100 mg/l + Vancomycin 50 mg/l + Phytagel 3g/l | 5,6 |
Có thể bạn quan tâm!
-
 Cấu Tạo Và Mối Liên Hệ Của Các Đường Thuộc Họ Raffinose
Cấu Tạo Và Mối Liên Hệ Của Các Đường Thuộc Họ Raffinose -
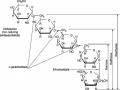
![Quá Trình Hoạt Động Của Cơ Chế Crispr/cas Ở Vi Khuẩn Chống Lại Sự Xâm Nhập Của Dna Ngoại Lai [51].](data:image/svg+xml,%3Csvg%20xmlns=%22http://www.w3.org/2000/svg%22%20viewBox=%220%200%2075%2075%22%3E%3C/svg%3E) Quá Trình Hoạt Động Của Cơ Chế Crispr/cas Ở Vi Khuẩn Chống Lại Sự Xâm Nhập Của Dna Ngoại Lai [51].
Quá Trình Hoạt Động Của Cơ Chế Crispr/cas Ở Vi Khuẩn Chống Lại Sự Xâm Nhập Của Dna Ngoại Lai [51]. -
 Nghiên Cứu Biểu Hiện Gen Và Chỉnh Sửa Hệ Gen Trên Rễ Tơ Đậu Tương
Nghiên Cứu Biểu Hiện Gen Và Chỉnh Sửa Hệ Gen Trên Rễ Tơ Đậu Tương -
 Sơ Đồ Minh Họa Cấu Trúc Vector Chỉnh Sửa Gen Crispr/cas9 Được Thế Kế Để Chuyển Gen Vào Đậu Tương
Sơ Đồ Minh Họa Cấu Trúc Vector Chỉnh Sửa Gen Crispr/cas9 Được Thế Kế Để Chuyển Gen Vào Đậu Tương -
 Kiểm Tra Hoạt Động Của Cấu Trúc Chỉnh Sửa Gen Crispr/cas9 Trên Các Dòng Rễ Tơ
Kiểm Tra Hoạt Động Của Cấu Trúc Chỉnh Sửa Gen Crispr/cas9 Trên Các Dòng Rễ Tơ -
 Sàng Lọc Các Dòng Đậu Tương Chỉnh Sửa Gen Qua Các Thế Hệ T1 Và T2
Sàng Lọc Các Dòng Đậu Tương Chỉnh Sửa Gen Qua Các Thế Hệ T1 Và T2
Xem toàn bộ 122 trang tài liệu này.
- Tạo nguyên liệu chuyển gen: hạt đậu tương chín sau khi được khử trùng, nảy mầm 3 ngày trên môi trường GCM, tách, thu phần lá mầm (tương tự như tạo nguyên liệu nuôi cấy rễ tơ in vitro ) và gây tổn thương bằng mũi dao nhọn từ 7 - 8 lần vào phần nốt lá mầm. Lá mầm đã được làm tổn thương sẽ dùng cho bước nhiễm khuẩn tiếp theo.
- Tạo dịch huyền phù vi khuẩn A. tumefaciens
Nuôi cấy tạo khuẩn lạc: cấy trải chủng vi khuẩn A. tumefaciens AGL1 mang cấu trúc chỉnh sửa hệ gen CRISPR/Cas9 từ ống giữ chủng lên môi trường YEP đặc, có bổ sung kháng sinh chọn lọc, nuôi trong tủ ổn nhiệt 28oC trong thời gian 48 giờ.
Nuôi lỏng khuẩn: dùng que cấy chọn một khuẩn lạc riêng biệt trên đĩa khuẩn cấy vào môi trường YEP lỏng bổ sung các loại kháng sinh như nuôi đặc. Bình nuôi lỏng được lắc 200 v/p ở 28oC trong 16 giờ ở điều kiện tối hoàn toàn trên máy lắc.
Tạo dịch huyền phù vi khuẩn: dịch khuẩn thu từ quá trình nuôi lỏng được hòa loãng 2 - 3 lần và tiếp tục nuôi phục hồi 2 - 4 giờ. Sau đó dịch khuẩn được ly tâm ở 5000 v/p trong 10 phút ở 4oC. Loại bỏ phần dịch nổi và hòa tan cặn khuẩn trong môi trường CCM lỏng có bổ sung AS và pha loãng cho đến khi dịch khuẩn có OD660 đạt 0,8 - 1. Dịch huyền phù này được dùng cho biến nạp.
- Nhiễm khuẩn và đồng nuôi cấy
Môi trường đồng nuôi cấy là CCM đặc có bổ sung AS. CCM được đổ trên đĩa petri, khi khô đặt giấy thấm đã khử trùng lên trên bề mặt môi trường. Mẫu được ngâm trong dịch huyền phù vi khuẩn trong thời gian 30 phút. Sau thời gian nhiễm khuẩn, mẫu được chuyển sang môi trường đồng nuôi cấy CCM đặc. Quá trình đồng nuôi cấy diễn ra trong tối, ở 25oC trong thời gian là 5 ngày.
- Diệt khuẩn và tạo đa chồi
Mẫu biến nạp sau thời gian đồng nuôi cấy được lắc trong môi trường SIM lỏng có bổ sung 500 mg/l cefotaxime với thời gian là 10 phút, sau đó thấm khô bằng giấy thấm khử trùng. Dùng panh và dao cắt bỏ chồi chính xuất hiện trên các mảnh lá mầm, cấy mẫu lên môi trường tạo cụm chồi có bổ sung 500 mg/l cefotaxime. Sau thời gian 2 tuần, mẫu được chuyển sang môi trường SIM đặc có bổ sung 500 mg/l cefotaxime, 3 mg/l PPT và nuôi trong 2 tuần.
- Tái sinh cây hoàn chỉnh
Các cụm chồi sống được trên môi trường chọn lọc sẽ được chuyển sang môi
trường phát triển chồi SEM có bổ sung 500 mg/l cefotaxime và 3 mg/l PPT. Khi các chồi phát triển đạt kích thước từ 3 - 5 cm sẽ được chuyển sang môi trường ra rễ RM có bổ sung 250 mg/l cefotaxime để tạo cây hoàn chỉnh.
- Kiểm tra tính kháng thuốc trừ cỏ
Các cây đậu tương chuyển gen thế hệ T0 tái sinh trên môi trường chọn lọc được chuyển ra trồng trên giá thể và sàng lọc bằng phết thuốc trừ cỏ (200 mg/l glufosinate) trên lá có ba lá chét. Sàng lọc bằng phết thuốc trừ cỏ trên lá được lặp lại 3 lần tại các vị trí khác nhau trên mỗi cây tái sinh.
2.3.6. Phương pháp trồng và chăm sóc cây đậu tương trong điều kiện nhà lưới
Cây đậu tương được trồng trong các chậu nhựa (chiều cao 25 cm, đường kính 20 cm) chứa hỗn hợp TRiBAT (Công ty TNHH MTV Sài Gòn xanh, Việt Nam) và trồng trong điều kiện nhà kính ở 28-35°C với quang chu kỳ 16 giờ sáng/8 giờ tối. Cây đậu tương được bón phân hai lần với NPK (15:5:20) sau khi trồng 40 ngày và NPK (16:16:16) sau khi trồng 65 ngày. Hạt đậu tương được thu hoạch và bảo quản trong kho lạnh (độ ẩm 40%, 4oC) cho các thí nghiệm tiếp theo. Các chỉ số sinh trưởng bao gồm chiều cao cây (cm), số nhánh, số lóng của mỗi cây được ghi lại khi cây ở giai đoạn trưởng thành (giai đoạn R8). Chiều dài lá và chiều rộng lá (cm) được đo tại các vị trí lá khác nhau ở giai đoạn phát triển R2 của cây
2.3.7 Phân tích sự di truyền của đột biến định hướng và gen chuyển các dòng đậu tương chỉnh sửa gen
Kiểm tra sự tính kháng thuốc trừ cỏ của dòng đột biến
Các dòng đậu tương T0, T1, T2 được kiểm tra sự có mặt của gen chọn lọc bar thông qua phương pháp phết thuốc trừ cỏ như mục 3.2.5. Lá của các dòng đậu tương chuyển gen ở các thế hệ khác nhau được sử dụng để tách chiết DNA tổng số theo phương pháp CTAB của Doyle và cộng sự (1991) và được sử dụng cho các phân tích tiếp theo.
Kiểm tra sự có mặt của gen chuyển
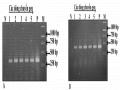
Các dòng đậu tương T1, T2 được kiểm tra sự có mặt của gen chuyển thông qua phản ứng PCR với cặp mồi đặc hiệu cho gen pcoCas9 và gen bar (Bảng phụ lục 1), với 35 chu kỳ khuếch đại. Sản phẩm PCR của các dòng đột biến được điện di trên gel agarose 1% để kiểm tra sự có mặt của gen chuyển.
Kiểm tra sự di truyền và phân ly của các đột biến trên các dòng đậu tương chỉnh sửa gen
DNA của các dòng đột biến thế hệ T1, T2 được sử dụng cho phân tích biến tính hồi tính trên gel polyacrylamide 15% theo phương pháp của Zhou và cộng sự (2014) [96] như trình bày ở mục 2.3.4. Sản phẩm PCR cũng được tinh sạch, tách dòng trong vector pJET1.2 (Thermo Scientific, Waltham, MA, USA) và giải trình tự bằng hệ thống ABI PRISM® 3100 Avant Genetic Analyzer tại tại Phòng thí nghiệm trọng điểm Công nghệ gen – Viện Công nghệ sinh học – Viện Hàn lâm khoa học và Công nghệ Việt Nam.
Xây dựng các chỉ thị phân tử để phát hiện các đột biến mất đoạn có kích thước nhỏ ở thế hệ T2 giống Maverick
Các chỉ thị phân tử được sử dụng cho việc sàng lọc và kiểm tra sự di truyền của các dòng đột biến phân ở thế hệ T2, xây dựng dựa trên nguyên tắc bổ sung với sợi khuôn DNA như Hình 2.2A, đồng thời kết hợp với kết quả giải trình tự của đột biến các gen GmGOLS ở thế hệ T1 của giống Maverick theo phương pháp của Đỗ Tiến Phát và cộng sự (2019) [87].
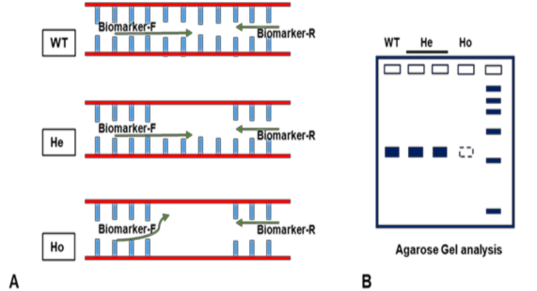
Hình 2.2. Sơ đồ minh họa nguyên lý thiết kế các chỉ thị phân tử dùng trong sàng lọc đột biến phân ly
A. Minh hoạ các chỉ thị phân tử bám được vào trình tự gen và không bám được vào trình tự gen do đột biến; B.Minh họa sản phẩm PCR trên bản gel agarose, ô màu thể hiện sự xuất hiện băng vạch trên bản gel, và ô nét đứt thể hiện sự không xuất
hiện băng vạch tương ứng trên bản gel
Kí hiệu và trình tự các chỉ thị phân tử như sau:
GOLS-seg F (5’-TGGAGTCACACCCCTCAGTA-3’), Tm: 560C; GOLS-seg F1 (5’-AGACATGGAGTCACACCCCG-3’), Tm: 600C; GOLS-seg F3 (5’-AGACATGGAGTCACACATGC-3’), Tm: 560C.
Khi kết hợp các chỉ thị phân tử này với các mồi đặc hiệu cho gen GmGOLS
G03R (5’- CCCCGTATATCTCCATGGCTTGG-3’); Tm: 560C G19R (5’- GCGCCAGAGCATGGCAAGGAC-3’); Tm: 560C
Trong đó, GOLS-seg F được thiết kế đặc hiệu để nhận biết alen WT (tức là alen không đột biến) và alen đột biến mất 22 bp (Δ-22 bp); GOLS-seg F1 được thiết kế để nhận biết alen mang đột biến Δ-33 bp; và GOLS-seg F3 được thiết kế để nhận biết alen mang đột biến Δ-30 bp. Khi chỉ thị phân tử kết hợp với một mồi đặc hiệu cho gen GmGOLS03hoặc GmGOLS19, thông qua PCR, các alen được khuếch đại và xuất hiện băng vạch khi điện di trên gel agarose (Hình 2.2 B).
Bảng 2.3. Dự kiến khả năng phát hiện các alen đột biến thông qua phản ứng PCR sử dụng các chỉ thị phân tử
GmGOLS03 | GmGOLS19 | ||||||
-22 | -33 | -22/-33 | -30 | WT | -22 | WT | |
GOLS-seg F + G03 R (Tm: 560C) | + | + | + | ||||
GOLS-seg F + G19 R (Tm: 560C) | + | + | |||||
GOLS-seg F1 + G03 R (Tm: 600C) | + | + | + | ||||
GOLS-seg F1 + G19 R (Tm: 600C) | |||||||
GOLS-seg F3 + G03 R (Tm: 560C) | + | + |
Ghi chú: (+) Kết quả dương tính với phản ứng PCR; WT. Alen không mang đột biến
Phương thức tiến hành như sau: từ các mẫu DNA của dòng đậu tương mang gen đột biến thế hệ T2, vùng gen GmGOLS03 và GmGOLS19 được khuếch đại bằng các chỉ thị phân tử với thành phần phản ứng bao gồm: 7,5µl DreamTaq green mastermix 2X (Thermo Scientific, Mỹ), 5 pM mồi xuôi, 5 pM mồi ngược, 100 ng DNA và 6,5 µl deion -H2O (đã khử DNAse và RNAse). Chu trình khuếch đại được thực hiện như sau: biến tính khởi đầu ở 94ºC trong 3 phút, khuếch đại trong 35 chu kỳ theo các bước lần lượt sau (1) biến tính ở 94ºC trong 30 giây, gắn mồi ở Tm (nhiệt độ gắn mồi) tùy thuộc chỉ thị phân tử trong 30 giây, (2) kéo dài ở 72ºC trong 30 giây,
(3) kéo dài kết thúc ở 72ºC trong 7 phút. Sản phẩm PCR được điện di trên gel agarose 2 %, các băng vạch DNA xuất hiện sẽ được dùng trong đánh giá sự di truyền của đột biến và hiệu quả của các chỉ thị phân tử.
Kết hợp kết quả xác định trình tự đột biến các gen GmGOLS ở thế hệ cây T1 và kết quả di truyền và phân ly của các đột biến thông qua phân tích PCR dự đoán sẽ thu được như Bảng 3.4.
2.3.8. Kiểm tra độ nảy mầm của hạt
Kiểm tra độ nảy mầm của hạt được thực hiện theo phương pháp xử lý nước của Blöchl và cộng sự (2007) [18]với các hạt phân ly ở thế hệ hệ T2 của cây mang đột biến đơn gen GmGOLS03 (gồm các dòng DT1.1-7-2, DT1.1-14-1, DT1.1-14-3) và đột biến kép hai gen GmGOLS03, GmGOLS19 (gồm các dòng DT1.1-5-3, DT1.1- 13-1, DT1,1-14-10), hạt ĐT26 không mang đột biến sử dụng làm đối chứng. Cụ thể hạt của các dòng mang đột biến gen có hàm lượng raffinose thấp và hạt ĐT 26 không mang đột biến (WT) được ngâm trong nước và theo dõi, so sánh tỉ lệ nảy mầm qua các mốc thời gian (24 giờ, 48 giờ, 72 giờ, 96 giờ và 120 giờ). Thí nghiệm lặp lại 3 lần cho mỗi dòng với 50 hạt mỗi lần lặp. Tốc độ nảy mầm được ghi lại sau mỗi 24 giờ cho đến 120 giờ, tính từ lúc thực hiện nảy mầm hạt.
2.3.9. Xác định thành phần hạt
Hạt đậu tương thế hệ T3 thu được từ các dòng T2 trồng trong nhà kính được nghiền và tiến hành định lượng tại viện nghiên cứu IPK, Đức:
- Phân tích hàm lượng ban đầu RFO tổng số từ các thành phần (Raffinose/ Sucrose / D-Glucose) trong hạt: thủy phân từng phần tạo các dạng đường đơn giản, sau đó phân tích chúng bằng bộ Megazyme Raffinose / Sucrose / D-Glucose (Megazyme Co, Bray, Ireland) theo hướng dẫn của nhà sản xuất.
- Phân tích về hàm lượng từng loại đường hòa tan bằng sắc ký lỏng cao áp (ICS- 3000, Thermo-Fisher). Các mẫu hạt T3 trưởng thành được nghiền và chiết bằng metanol (80% v/v) theo phương pháp của Weber và cộng sự (2000). Việc tách chiết mẫu được thực hiện trên cột PA1 (2 × 250 mm) và cột bảo vệ (PA1 2 × 50 mm) ở 40°C bằng cách chạy đẳng tích với 100 mM NaOH ở tốc độ dòng không đổi là 0,35 ml/phút. Thành phần các loại đường sẽ được xác định căn cứ vào thời gian lưu giữ tương ứng với các tiêu chuẩn đã cài đặt trước đó. Hiệu chuẩn bên ngoài đã được áp dụng bằng cách sử dụng các tiêu chuẩn đã xác thực.
- Phân tích hàm lượng chất béo, tinh bột, protein và độ ẩm của hạt bằng máy quang phổ kế (MPA; Bruker), được hiệu chuẩn theo quy trình của nhà cung cấp.
2.3.10. Phân tích các đột biến ngoài mục tiêu (off target)
DNA của các dòng đậu tương T2 từ cùng cây T1 có hàm lượng cacbohydrate thay đổi được nhóm lại với nhau và kiểm tra hiện tượng off-target có thể xảy ra. Các off-target được dự đoán bởi chương trình dự đoán trực tuyến CCTop theo phương pháp của Stemmer và cộng sự (2015). Sản phẩm PCR của các dòng đột biến được tách dòng và giải trình tự gen theo phương pháp Sanger như trình bày bên trên. Việc giải trình tự được thực hiện tại Phòng thí nghiệm trọng điểm Công nghệ gen – Viện Công nghệ sinh học – Viện Hàn lâm khoa học và Công nghệ Việt Nam.
2.3.11. Phân tích thống kê
Các số liệu được phân tích bởi SPSS (version 16.0) và Microsoft Excel 2007 (Microsoft, Redmond, WA, Hoa Kỳ). ANOVA một chiều được sử dụng để phân tích các nhóm đột biến và t-test để phân tích các giá trị trung bình những dòng đột biến đơn; với mức ý nghĩa thống kê ở p = 0,05. Đối với phân tích thành phần hạt giống HPLC, mỗi kiểu gen đột biến gây ra được biểu thị bằng hạt từ hai dòng; phép đo của mỗi dòng được thực hiện trong bốn lần.
CHƯƠNG 3
KẾT QUẢ NGHIÊN CỨU
3.1 Thiết kế hệ thống chỉnh sửa CRISPR/Cas9 nhằm gây tạo đột biến các gen
GmGOLS trên đậu tương
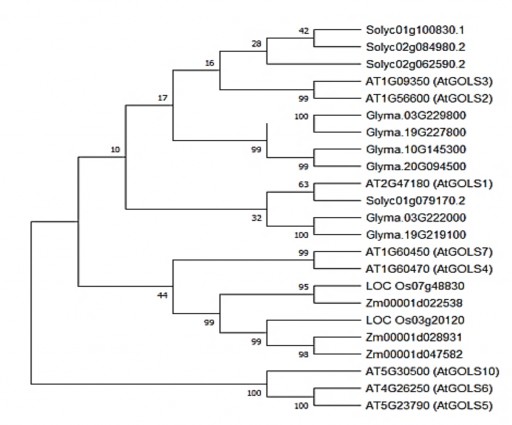
Lựa chọn trình tự gen mã hóa Galactinol synthase (GmGOLS) và thiết kế cấu trúc chỉnh sửa hệ gen CRISPR/Cas9. Thông qua công cụ tìm kiếm BLAST, chúng tôi đã phân tích, kiểm tra mối quan hệ phát sinh loài giữa các GmGOLS đậu tương và các loài thực vật khác. Dựa trên trình tự protein Arabidopsis GOLS, 6 gen GmGOLS giả định đã được xác định từ cơ sở dữ liệu hệ gen của đậu tương SoyBase.org (Hình 3.1).
Hình 3.1. Mối quan hệ phát sinh loài giữa các protein GOLS đã được xác định trong cây Arabidopsis, cà chua, ngô 23 trình tự protein GOLS
(đã được sử dụng để xây dựng cây phát sinh loài bằng phương pháp phân tích Neighbor Joining, sử dụng phần mềm MEGA X với 1000 lần lấy mẫu)
Đồng thời, tham khảo cơ sở dữ liệu về mức độ phiên mã của các gen GmGOLS khác nhau trong các mô khác nhau và ở các giai đoạn phát triển khác nhau của hạt ở Hình 3.2, chúng tôi nhận thấy có sáu gen GmGOLS (Glyma.03G222000,

![Quá Trình Hoạt Động Của Cơ Chế Crispr/cas Ở Vi Khuẩn Chống Lại Sự Xâm Nhập Của Dna Ngoại Lai [51].](https://tailieuthamkhao.com/uploads/2023/02/20/nghien-cuu-ung-dung-cong-nghe-crispr-cas9-trong-tao-dot-bien-gen-gmgols03-5-1-120x90.jpg)