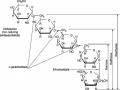
ngắn (được gọi là crRNA_CRISPR-derived RNA), các crRNA này sẽ liên kết với endonuclease Cas để nhận diện DNA mục tiêu (thông qua liên kết bổ sung của trình tự crRNA và DNA mục tiêu) và cắt phân tử DNA mục tiêu (thông qua hoạt động endonuclease của Cas) [48] (Hình 1.5).
CRISPR/Cas được phân nhóm dựa vào sự tham gia của nuclease Cas và cấu trúc đoạn lặp lại trong CRISPR. Trong đó, phổ biến nhất là CRISPR loại II có sự tham gia của một endonuclease mang vùng tương đồng với enzyme resolvase (RuvC) và vùng Histidine-Asparagine-Histidine (H-N-H domain), được gọi là Cas9 (CRISPR associated protein 9) [49]. Hệ thống CRISPR/Cas9 được ứng dụng phổ biến trong các nghiên cứu chỉnh sửa gene và cải thiện cây trồng [50].
Hình 1.5. Quá trình hoạt động của cơ chế CRISPR/Cas ở vi khuẩn chống lại sự xâm nhập của DNA ngoại lai [51].
1.3.2. Cơ chế hoạt động của hệ thống chỉnh sửa gen CRISRP/Cas9
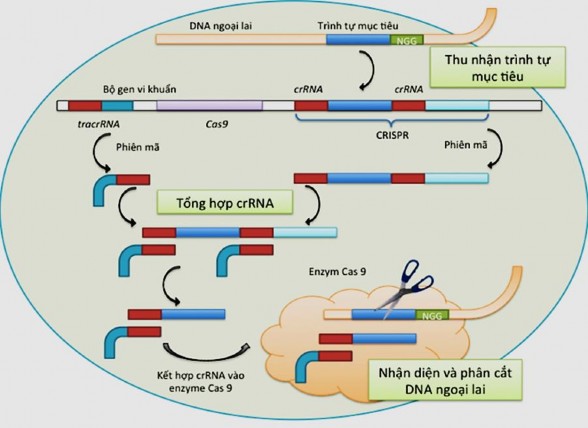
Với hệ CRISPR/Cas9, các trình tự CRISPR và vùng đệm được phiên mã thành crRNA (CRISPR-RNA). Sau đó phân tử crRNA này được cắt lọc một số phần và hoàn thiện cấu trúc tạo thành phân tử tracrRNA đầy đủ chức năng dẫn đường cho Cas9 đến cắt DNA ở vị trí xác định. Quá trình biến đổi này được xúc tác bởi enzyme
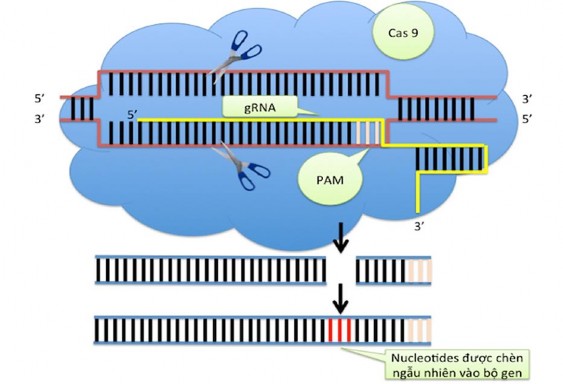
RNase III. crRNA chứa một trình tự phiên mã của CRISPR và DNA ngoại lai. Nó đóng vai trò nhận biết chính xác vị trí cắt trên trình tự của DNA ngoại lai. Sự kết hợp giữ crRNA bám vào tracrRNA tạo ra một phức hệ thông minh (crRNA/tracrRNA hay hay guide RNA) vừa có thể cho Cas9 biết chính xác vị trí và thực hiện cắt phân tử DNA ngoại lai tại vị trí đó [52]. Tuy nhiên, để Cas9 có thể bám vào sợi xoắn kép DNA ngoài sự có mặt của crRNA cần phải có thêm sự hiện diện của một motif nằm cạnh tiền vùng đệm (proto-spacer adjacent motif) hay viết tắt là PAM. Cas9 chỉ có thể bám vào sợi xoắn kép DNA nếu một trình tự PAM có ba nucleotide có chứa hai guanine và một base bất kỳ khác nằm cạnh trình tự nhận diện. Khi phức hệ Cas9 và gRNA được hình thành và bám vào vùng định hướng của DNA ngoại lai, hai mạch của DNA bị tháo xoắn và phân tử crRNA/tracrRN gắn bổ sung với trình tự định hướng (target) trên DNA sợi đơn. Protein Cas9 sau đó thực hiện cắt đặc hiệu tại hai sợi đơn DNA ở cùng một vị trí cách trình tự PAM từ 2-3 nucleotides [53], Hình 1.6
Có thể bạn quan tâm!
-
 Nghiên cứu ứng dụng công nghệ CRISPR/Cas9 trong tạo đột biến gen GmGOLS03, GmGOLS19 trên cây đậu tương Glycine max L. Merrill nhằm giảm lượng đường họ Raffinose trong hạt - 2
Nghiên cứu ứng dụng công nghệ CRISPR/Cas9 trong tạo đột biến gen GmGOLS03, GmGOLS19 trên cây đậu tương Glycine max L. Merrill nhằm giảm lượng đường họ Raffinose trong hạt - 2 -
 Tình Hình Sản Xuất Và Chọn Tạo Giống Đậu Tương
Tình Hình Sản Xuất Và Chọn Tạo Giống Đậu Tương -
 Cấu Tạo Và Mối Liên Hệ Của Các Đường Thuộc Họ Raffinose
Cấu Tạo Và Mối Liên Hệ Của Các Đường Thuộc Họ Raffinose -
 Nghiên Cứu Biểu Hiện Gen Và Chỉnh Sửa Hệ Gen Trên Rễ Tơ Đậu Tương
Nghiên Cứu Biểu Hiện Gen Và Chỉnh Sửa Hệ Gen Trên Rễ Tơ Đậu Tương -
 Chuyển Gen Tạo Cây Đậu Tương Mang Cấu Trúc Crispr/cas9
Chuyển Gen Tạo Cây Đậu Tương Mang Cấu Trúc Crispr/cas9 -
 Sơ Đồ Minh Họa Cấu Trúc Vector Chỉnh Sửa Gen Crispr/cas9 Được Thế Kế Để Chuyển Gen Vào Đậu Tương
Sơ Đồ Minh Họa Cấu Trúc Vector Chỉnh Sửa Gen Crispr/cas9 Được Thế Kế Để Chuyển Gen Vào Đậu Tương
Xem toàn bộ 122 trang tài liệu này.
Hình 1.6. Cơ chế hoạt động của CRISPR/Cas9 trong trường hợp gây biến đổi trình tự gen [51]
Nếu có một đoạn DNA mẫu (template DNA, hay donor DNA) tương đồng với vị trí mục tiêu thì DNA sợi đôi bị đứt gãy này có thể được sửa chữa bằng cơ chế HDR. Phần DNA ở quanh vị trí đứt gãy sẽ được thay thế bằng phần DNA tương ứng ở DNA mẫu. Nếu một đột biến được chủ ý đưa vào DNA mẫu thì đoạn DNA sau khi
sửa chữa sẽ mang đột biến đó (Hình 1.7A) [51].
Khi mẫu DNA có số lượng lớn cơ chế chỉnh sửa theo hướng HDR và NHEJ, có thể cùng xảy ra đồng thời dẫn tới hiện tượng chèn/xóa đoạn không mong muốn. Khi đó biến thể của Cas9 là Cas9D10A sẽ hạn chế điều này. Cas9D10A khác với Cas9 thông thường là nó chỉ có khả năng cắt trên một mạch DNA thay vì hai mạch, do đó không kích hoạt cơ chế NHEJ mà việc sửa chữa DNA chỉ được tiến hành theo cơ chế HDR [54]. Nếu sử dụng từ hai phức hợp Cas9D10A để tạo nên các vị trí đứt gãy sợi đơn (nick) cạnh nhau sẽ làm tăng sự đặc hiệu mục tiêu [55] (Hình 1.7)
Biến thể thứ ba của Cas9 là nuclease-deficient Cas9 (dCas9) (Hình 1.7C) không có khả năng cắt DNA). Các đột biến xảy ra với HNH và RuvC domain bất hoạt khả năng cắt DNA, nhưng vẫn giữ lại khả năng liên kết với DNA. Ứng dụng của biến thể này là biến dCas9 thành công cụ hoạt hóa hoặc ức chế phiên mã khi dung hợp dCas9 với các domain tác động (hoạt hóa hoặc ức chế). Ngoài ra, có thể lợi dụng khả năng bắt cặp đặc hiệu trình tự của dCas9 với DNA để hiển thị trình tự DNA mong muốn trên genome bằng cách dung hợp dCas9 với protein huỳnh quang [56]. Nhờ những biến thể trên CRISPR/Cas9 ngày càng được sử dụng rộng trong việc tạo ra thực vật biến đổi gen với hiệu quả cao.
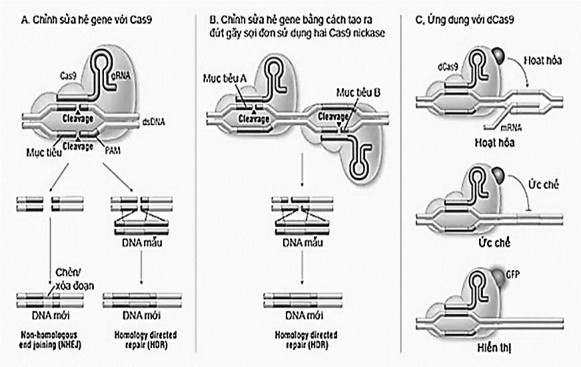
Hình 1.7. Chỉnh sửa hệ gene bằng hệ thống CRISPR-Cas9 (Nguồn: [51])
1.3.3. Ứng dụng hệ thống CRISPR/Cas9 trong chọn tạo giống ở đậu tương
Chỉnh sửa hệ gen là kỹ thuật tạo ra những thay đổi trên trình tự gen nội sinh theo cách có chủ đích tại một vị trí xác định khác với đột biến gen. Công nghệ chỉnh sửa gen tạo ra các đặc tính ưu việt cho cây trồng. Đến nay có 24 đối tượng cây trồng, với ít nhất 193 gen đã được chỉnh sửa bằng hệ thống này [57].
Kanazashi đã chuyển gen thông qua Agrobacterium sử dụng một sgRNA gây đột biến định hướng đồng thời hai locus GmPPD trong đậu tương. Các đột biến ở locus GmPPD được xác nhận trong ít nhất 33% hạt T2 [58]. Ngoài ra, trên cùng một gen mục tiêu, sử dụng cùng một cấu trúc CRISPR/Cas9 nhưng các sgRNA khác nhau có thể dẫn đến hiệu quả đột biến khác nhau [59].
Nhóm nghiên cứu của Mengyan Bai đã khai thác 74 gen liên quan đến việc hình thành nốt sần ở rễ và 28 gen chức năng có trong hạt đậu tương để cải tạo di truyền, xây dựng quần thể đột biến gen mục tiêu [60]. Jie Wang và cộng sự đã thành công trong việc tạo giống đậu tương cải biến gen tạo hạt không chứa lipoxygenase (chất tạo mùi đặc trưng) được qui định bởi ba gen (LOX1, LOX2, LOX3). Hệ thống CRISPR/Cas9 được thiết kế nhắm mục tiêu vào ba gen GmLox (GmLox1, GmLox2 và GmLox3) đã được áp dụng, 60 cây chuyển gen T0 dương tính được tạo ra, mang các tổ hợp của sgRNA và đột biến. Thử nghiệm so màu cho thấy rằng các cây mang các tổ hợp đột biến khác nhau đã mất các hoạt động lipoxygenase tương ứng. Các đột biến không chuyển gen thu được bằng cách sàng lọc thế hệ T2 của các dòng đột biến không có lipoxygenase (GmLox-28 và GmLox-60) [61].
Công nghệ CRISPR/Cas9 được sử dụng để tạo ra các vector loại trực tiếp đột biến đơn và kép các gen chức năng mã hóa enzyme sinh tổng hợp axit béo họ FAD2. Xác định trình tự của gen mục tiêu thể hiện hiệu suất chỉnh sửa gen GmFAD2-03 và GmFAD2-2A lần lượt là 95% và 55,56%, hiệu suất chỉnh sửa đột biến kép là 66,67%. Kết quả ở thế hệ T2, hạt đậu tương có hàm lượng axit oleic tăng từ 17,10% lên 73,50%; hàm lượng axit linoleic giảm từ 62,91% xuống 12,23%; hàm lượng protein tăng từ 37,69% lên 41,16% [62].
CRISPR/Cas9 được Peipei Zhang và cộng sự ứng dụng trong nghiên cứu chỉnh sửa đa gen của cây đậu tương (GmF3H1, GmF3H2 và GmFNSI ‐1) nhằm cải thiện hàm lượng isoflavone và khả năng kháng virus khảm trên cây. Phân tích chuyển hóa của lá đột biến thế hệ T0 cho thấy hàm lượng isoflavone cải thiện đáng kể [63].
Zhaobo Liab và cộng sự đã thành công trong nghiên cứu ứng dụng CRISPR/Cas9 gây đột biến có mục tiêu bốn gen LNK2 có nguồn gốc từ cây Arabidopsis thaliana nhằm làm tăng thời gian ra hoa của đậu tương, kết quả thể đột biến tứ bội ra hoa sớm hơn đáng kể so với loại dại (WT) [64].
Zhandong Cai và cộng sự đã tiến hành thí nghiệm loại bỏ gen GmJAG1 bằng kỹ thuật CRISPR/Cas9, kết quả đã làm tăng năng suất giống đậu tương Huachun 6 trồng ở khu vực có vĩ độ thấp, năng suất của các dòng đột biến đã tăng lần lượt là 8,81% và 8,67% hơn giống đối chứng trong thử nghiệm mùa xuân và mùa hè [65].
Các dòng bất dục đực tế bào chất của đậu tương đã được sử dụng trong các hệ thống nhân giống lai ba dòng, tuy nhiên về ưu thế lai của đậu tương bị hạn chế bởi nguồn gen hiếm của các dòng bất dục. Bằng công nghệ CRISPR/Cas9, Xiao Chen và cộng sự đã chỉnh sửa mục tiêu tác động đến việc tổng hợp các chất tương đồng AMS có trong đậu tương để tạo ra các dòng bất dục đực ổn định. Việc chỉnh sửa có mục tiêu GmAMS1 dẫn đến kiểu hình bất dục đực, trong khi chỉnh sửa GmAMS2 không tạo ra dòng bất dục đực. GmAMS1 không chỉ có chức năng trong việc hình thành màng phấn mà còn kiểm soát sự thoái hóa của tapetum đậu tương [66].
Hình 1.8. Chỉnh sửa gen bằng hệ thống CRISPR/Cas9 trên một số loại cây trồng (giai đoạn tháng 8/2013-8/2018) [57].
1.3.4 Phương pháp đánh giá hoạt động của cấu trúc chỉnh sửa hệ gen
Có nhiều phương pháp dùng để đánh giá hoạt động của cấu trúc chỉnh sửa hệ gen như hệ thống biểu hiện tạm thời, sử dụng tế bào trần, hệ thống nuôi cấy rễ tơ. Tuy nhiên, tùy thuộc vào từng đối tượng nghiên cứu mà các nhà khoa học lựa chọn các phương pháp tối ưu.
1.3.4.1 Đánh giá hiệu quả hoạt đồng của hệ thống CRISPR/Cas9 qua biểu hiện tạm thời
Bước quan trọng trong nghiên cứu chỉnh sửa hệ gen là đánh giá hoạt động của hệ thống vector chỉnh sửa gen CRISPR/Cas9. Các nghiên cứu đầu tiên đã sử dụng hệ thống biểu hiện tạm thời trên lá để đánh giá hoạt động của các hệ thống CRISPR/Cas9 đã được thiết kế. Cụ thể các nhà khoa học đột biến có chủ đích đồng thời ba gen ở lúa mì bằng kỹ thuật CRISPR/Cas9, đồng thời họ thiết kế một phương pháp phát hiện nhanh đột biến thông qua sự biểu hiện lưỡng cực của gRNA và Cas9 có trong phôi lúa mì chưa trưởng thành. Sau đó các dạng đột biến được xác định bằng kỹ thuật PCR-RFLP ở tuần kế tiếp [67]. Hay sự biểu hiện tạm thời trên lá cây ca cao mang gen chuyển TcNPR3 biểu hiện kháng bệnh Phytophthora Tropicalis được chuyển vào thông qua hệ thống CRISPR/Cas9, các đột biến được phát hiện thông qua việc giải trình gen [68].
1.3.4.2 Đánh giá hiệu quả hoạt động của hệ thống CRISPR/Cas9 sử dụng tế bào trần
Bên cạnh hệ thống biểu hiện tạm thời trên lá, hệ thống nuôi cấy tế bào trần cũng được ứng dụng nhiều trong đánh giá hoạt động của hệ thống chỉnh sửa hệ gen trên nhiều đối tượng cây trồng khác nhau thể hiện qua các công trình nghiên cứu như: Junpin Gao và cộng sự đã gây đột biến có mục tiêu hai gen (NtPDS và NtPDR6) ở thuốc lá bằng kỹ thuật CRISPR/Cas9, sau đó họ kiểm tra nhanh hoạt động của hệ thống chỉnh sửa bộ gen này trong nguyên bào thuốc lá, đột biến chèn và xóa (indel) được ghi nhận với tần số từ 16,2% đến 20,3% sau khi truyền RNA dẫn đường (gRNA) và nuclease Cas9 trong nguyên bào thuốc lá. Kết quả cuối của nghiên cứu nhóm thu được cây thuốc lá chuyển gen với đột biến NtPDS và NtPDR6 do Cas9/gRNA gây ra. Tỉ lệ đột biến là 81,8% đối với NtPDS gRNA4 và 87,5% đối với NtPDR6 gRNA2, không thu được đột biến đáng kể ngoài mục tiêu [69]. Việc đánh giá hoạt động của cấu trúc chỉnh sửa hệ gen thông qua tế bào trần còn được áp dụng trên cây nho, táo
[70] và một số loại cây trồng khác [71].
1.3.4.3 Đánh giá hiệu quả hoạt động của hệ thống CRISPR/Cas9 thông qua hệ thống nuôi cấy rễ tơ
Bên cạnh hệ thống biểu hiện tạm thời và hệ thống nuôi cấy tế bào trần, gần đây hệ thống nuôi cấy rễ tơ đã được ứng dụng thành công trong đánh giá hoạt động của hệ thống chỉnh sửa gen trên một số đối tượng cây trồng. Điển hình trên cây lạc, CRISPR/Cas9 được áp dụng nhằm loại bỏ hai gen AhNFR1 và AhNFR5 tạo nốt sần. Thông phương pháp tạo rễ tơ nhóm nghiên cứu đã đánh giá được hiệu quả hoạt động của hệ thống CRISPR/Cas9 đồng thời xác định được chức năng của gen AhNFR5 trong sự hình thành nốt sần ở cây [72].
Thông qua hệ thống biểu hiện rễ tơ trên cây rau diếp xoắn, hệ thống CRISPR/Cas9 được đánh giá có hiệu quả trong việc loại bỏ gen CiPDS, tần số đột biến là 4,5% đối với tế bào trần, 31,25% với biến nạp qua trung gian A. rhizogenes với nhiều biallelic được phát hiện ở tất cả các cây mang đột biến [73]. Trên cây khoai tây hệ thống cảm ứng tạo rễ tơ được sử dụng để đánh giá hoạt động của hệ thống CRISPR/Cas9 trong chỉnh sửa gen StPDS mã hoá cho enzyme liên quan đến việc tổng hợp các carotenoid, kết quả 64% - 98% dòng rễ tơ chuyển gen mang đột biến mục tiêu được biểu hiện, 14% –30% đột biến dạng thể khảm. Các đột biến được duy trì trong các dòng tái sinh là các đột biến ổn định với tỉ lệ trung bình là 38% và có khả năng truyền dòng mầm cho thế hệ con cháu [74].
1.4. Hệ thống cảm ứng tạo rễ tơ và ứng dụng trong nghiên cứu biểu hiện gen và chỉnh sửa hệ gen trên đậu tương.
1.4.1. Hệ thống cảm ứng rễ tơ ở thực vật
Rễ tơ là một bệnh ở thực vật gây ra bởi quá trình tương tác giữa vi khuẩn A. rhizogenes, đây là một loại vi khuẩn đất gram âm - với tế bào vật chủ. Trong quá trình xâm nhiễm, vùng T-DNA ở giữa đoạn biên phải (right border) và đoạn biên trái (left border) trên Ri-plasmid trong vi khuẩn được chuyển và tích hợp vào genome của thực vật. Vùng T-DNA này có chứa các gen mã hóa cho các loại protein khác nhau, trong đó có protein tham gia vào quá trình sinh tổng hợp các hóc môn thực vật. Do vậy, biểu hiện của các gen này kích thích hình thành các rễ tơ tại hoặc gần vị trí xâm nhiễm. Sự cảm ứng rễ tơ được chứng minh ở nhiều cơ quan thực vật khác nhau, lá, cơ quan dự trữ và lá mầm cây non và mô thân.
Giống như Ti-plasmid ở vi khuẩn A. tumefaciens, Ri-plasmid (Root-inducing plasmid) ở vi khuẩn A. rhizogenes là phân tử DNA mạch vòng, sợi kép có khối lượng phân tử lớn từ 200-800 kb gồm 2 vùng chính là T-DNA và vir (virulence). Dựa vào khả năng sinh tổng hợp các opine ở rễ tơ, Ri-plasmid được chia làm 2 nhóm chính: agropine và mannopine, và một số nhóm phụ được tìm thấy và phân loại sau này như cucumopine và mikimopine. Trong đó, các chủng thuộc nhóm agropine (A4, 15834, LBA9402, 1855) được chứng minh là độc hơn so với chủng thuộc nhóm mannopine (8196, TR7, TR101) và các chủng này được sử dụng chủ yếu trong các nghiên cứu tạo rễ tơ ở thực vật [75]. Phần T-DNA của Ri-plasmid có chứa các gen tương đồng với các gen trên T-DNA của Ti-plasmid gây khối u trên vi khuẩn A. tumefaciens, ví dụ như gen có vai trò trong quá trình sinh tổng hợp agropine. Khi Ri-plasmid được chuyển vào các tế bào chủ thông qua tổn thương, T-DNA sẽ được chèn ổn định vào hệ gen của tế bào chủ [76]. Các gen mã hóa trên T-DNA có nguồn gốc từ vi khuẩn nhưng có các trình tự điều khiển của tế bào nhân chuẩn giúp cho việc biểu hiện ở các tế bào chủ bị lây nhiễm. Các gen này mã hóa cho các thành phần chức năng tham gia trong quá trình sinh tổng hợp auxin. Ngoài chúng cũng tham gia vào việc tổng hợp các hợp chất kích thích phân chia của tế bào để hình thành rễ tơ dưới ảnh hưởng của auxin nội sinh [77].
Chủng A. rhizogenes có khả năng xâm nhiễm qua vết thương của hầu hết các loài thực vật hai lá mầm và một số ít các loài thực vật một lá mầm. Hầu hết các vật liệu như trụ dưới lá mầm, lá, chồi đỉnh, cuống hoa, gân lá, đỉnh chồi, lá mầm, tế bào trần rễ dự trữ/củ có thể bị lây nhiễm khuẩn và cảm ứng để tạo rễ tơ [78]. Để cảm ứng hình thành rễ tơ, mẫu thực vật được làm tổn thương và đồng nuôi cấy hoặc ủ với vi khuẩn A.rhizogenes. Thông thường sau 2-3 ngày, mẫu thực vật được chuyển lên môi trường đặc có kháng sinh phù hợp như cefotaxime, carbecillin, vancomycin, ampicillin, claforan, streptomycin hoặc tetracycline với nồng độ dao động từ 100 đến 500µg/ml để diệt và loại bỏ vi khuẩn còn sót lại [78]. Kết quả của việc lây nhiễm là sự hình thành rễ tơ tại hoặc xung quanh vị trí mô tổn thương. T-DNA của Ri-plasmid có thể được thiết kế lại với các gen quan tâm và sử dụng trong công tác chuyển gen. Do mỗi rễ tơ tạo ra được xem như một dòng chuyển gen độc lập nên có thể thu được số lượng lớn dòng chuyển gen trong khoảng thời gian ngắn [79].
Hệ thống cảm ứng rễ tơ thông qua A. rhizogenes đang được ứng dụng rộng rãi