GmGOLS19của dòng M3.1, tất cả các vùng khác đều chứa hai hoặc nhiều alen đột biến cho thấy sự xuất hiện của các thể khảm, do đó đòi hỏi phải sàng lọc thêm thế hệ tiếp theo để xác định tính di truyền của các đột biến và lựa chọn các đột biến ổn định.
3.4 Sàng lọc các dòng đậu tương chỉnh sửa gen qua các thế hệ T1 và T2
Sau khi thu được các dòng cây đột biến và phân tích trình tự các đột biến nhận được trên 2 gen quan tâm, các dòng đậu tương mang đột biến này được chăm sóc trong điều kiện nhà lưới và tiến hành thu hạt ở các thể hệ khác nhau để đánh giá khả năng di truyền, tính phân ly của các đột biến định hướng. Do hai giống đậu tương ĐT26 và Mr có đặc điểm sinh trưởng và yêu cầu chăm sóc khác nhau thế nên các dòng cây thuộc hai giống này được tiến hành phân tích riêng.
3.4.1 Dòng DT 1.1 của giống ĐT26
3.4.1.1. Phân tích sự di truyền, phân ly của gen chọn lọc bar ở thế hệ T1
Hệ thống vector chuyển gen CRISPR/Cas9 được thiết kế dùng tạo đột biến có chứa gen chọn lọc bar nhằm bước đầu xác định các dòng cây mang gen chuyển. Thông qua phương pháp phết lá với thuốc diệt cỏ glufosinate nồng độ 200 mg/l, sau đó được xác nhận bằng sàng lọc PCR sự có mặt của trình tự T-DNA sử dụng cặp mồi đặc hiệu cho gen pcoCas9 và vùng mở rộng 35SPPDK – pFGC trong gen chuyển cụ thể tại Bảng phụ lục 1. Các dòng đậu tương thế hệ T1 được phết trực tiếp glufosinate 200 mg/l trên bề mặt lá.
Kết quả sau thí nghiệm thu được 29 cây đậu tương đột biến thế hệ T1 của dòng DT1.1 thuộc giống ĐT26 không mẫn cảm với glufosinate 200 mg/l, phân tích mẫu các cây T1 bằng phản ứng PCR với các cặp mồi đặc hiệu (Bảng phụ lục 1), sản phẩm PCR phân tích điện trên gel agarose 1% cho kết quả dương tính với gen bar. Kết hợp với kết quả sử dụng phương pháp phết lá với thuốc diệt cỏ (Bảng phụ lục 2) đã chứng minh gen chuyển bar được di truyền và phân ly ở các dòng đột biến thuộc thế hệ T1.
3.4.1.2 Sự di truyền, phân ly của đột biến các gen GmGOLS ở thế hệ T1
Các dòng T0 được trồng trong điều kiện nhà lưới và thu hạt để đánh giá sự di truyền của các alen GmGOLS đột biến ở thế hệ T1. Dựa vào kết quả điện di trên gel agarose đã xác định được sự phân ly của đột biến mất đoạn lớn (∆-23 bp và ∆-77 bp) trên gen GmGOLS03và GmGOLS19 ở thế hệ T1 của dòng chuyển gen DT1.1 (Hình 3.15A).
Đối với gen GmGOLS03, 7 trong số 15 cây T1 được kiểm tra cho thấy sự xuất
hiện của hai băng vạch; tuy nhiên tất cả đều thấp hơn băng vạch khuếch đại của cây đối chứng. Ngoài ra, bốn cây T1 chỉ có một băng vạch trên và những cây còn lại chỉ có một băng vạch dưới tương ứng kiểu gen đồng hợp tử với hai dạng đột biến này.
Đối với gen GmGOLS19, 8 cây T1 xuất hiện hai băng vạch, trong khi 5 cây khác chỉ mang một băng vạch dưới và hai cây còn lại mang một băng vạch trên (Hình 3.15A). Điều quan trọng là hai cây T1 (DT1.1-5 và DT1.1-6) chỉ có băng vạch dưới của cả hai gen GmGOLS03 và GmGOLS19, chứng tỏ các dòng này bị mất đoạn lớn ở cả hai gen GmGOLS.
Hình 3.15. Sự phân ly của các đột biến ở thế hệ T1 và T2 của dòng DT1.1
(A) và (B) lần lượt là kết quả điện di sản phẩm PCR của các trình tự đích trên gen GmGOLS03 và GmGOLS19 ở thế hệ T1 và T2. WT: cây đối chứng giống ĐT26;
DT1.1-1 đến DT1.1-15: cây T1 của dòng đột biến DT1.1; M: thang DNA 1 kb ở ảnh cây T1 và thang DNA 100 bp ở ảnh cây T2; DT1.1-5-1 đến DT1.1-5-6: cây T2 từ dòng DT1.1-5; DT1.1-7-1 đến DT1.1-7-5: cây T2 từ dòng DT1.1-7
Kết quả giải trình tự vùng gen quan tâm của các dòng T1 được lựa chọn (DT1.1-2, DT1.1-5, DT1.1-6, DT.1-7, DT1.1-13, DT1.1-14) cho thấy: các cây đột
biến gen phân ly ở thế hệ T1 thuộc các dòng DT1.1-5, DT1.1-6, DT1.1-13 đều mang đột biến kép ở cả hai gen GmGOLS, trong khi những dòng còn lại DT1.1-2, DT1.1-7 và DT1.1-14 là đột biến biallelic/đồng hợp tử trên gen GmGOLS03 và đột biến dị hợp tử trên gen GmGOLS19 (Hình 3.16). Kết quả giải trình tự vùng gen đích của GmGOLS03 ở thế hệ T1 tương thích với kết quả phân tích điện di trên gel agarose
(Hình 3.15A). Các dòng DT1.1-5, DT1.1-6 và DT1.1-14 mang đột biến mất đoạn ∆- 77 bp, trong khi DT1.1-7 và DT1.1-13 mang đột biến ∆-32 bp.
Hình 3.16. Kết quả phân tích sự phân ly của các đột biến trên gen GmGOLS GmGOLS03 (A) và GmGOLS19 (B) ở thế hệ T1 của các dòng DT1.1, M3.1 và M4.1. Vùng chỉnh sửa đích được đánh dấu màu đỏ (sgRNAs); vùng PAM được đánh dấu màu xanh; ∆ thể hiện sự thay đổi trong trình tự đích: không thay đổi (0), mất nucleotide (-),thêm nucleotide (+).
Ngoài ra, dòng DT1.1-2 chứa cả hai đột biến mất đoạn ∆-32 bp và ∆-77 bp (biallelic). Giải trình tự gen GmGOLS19 cho thấy các dòng T1 mang alen wild-type và alen ∆-23 bp; DT1.1-5, DT1.1-6, DT1.1-13 là dòng đột biến đồng hợp tử trong khi DT1.1-2, DT1.1-7 và DT1.1-14 là dòng đột biến dị hợp tử.
Như vậy, ở thế hệ T1 đối với giống ĐT26, đã thu được 03 dòng đột biến đồng hợp tử ở cả hai gen GmGOLS. Ngoài ra, không ghi nhận được alen wild-type nào của gen GmGOLS03 ở tất cả các dòng T1 được kiểm tra, mặc dù chúng xuất hiện ở thế hệ T0 (DT1.1) (Hình 3.14, Hình 3.16); điều này đồng nghĩa với việc không thu được dòng đột biến đơn gen GmGOLS19. Các đặc điểm kiểu gen và kiểu hình tiếp theo sẽ được thực hiện trên các dòng đột biến đơn GmGOLS03 và dòng đột biến kép GmGOLS03, GmGOLS19.
3.4.1.3 Sự di truyền, phân ly của thế hệ cây T2 của dòng DT 1.1 giống ĐT26
Sự di truyền ổn định của tất cả các alen đột biến trên gen GmGOLS03 và
GmGOLS19 của dòng DT1.1 tiếp tục được đánh giá ở các dòng thuộc thế hệ T2 (Hình
3.15 B, Hình3.17).
Hình 3.17. Sự phân ly của các đột biến trên gen GOLS ở thế hệ T2 của dòng DT1.1
(A) GmGOLS03 và (B) GmGOLS19
Vùng chỉnh sửa đích được đánh dấu màu đỏ (sgRNAs); vùng PAM được đánh dấu màu xanh; ∆ thể hiện sự thay đổi trong trình tự đích: không thay đổi (0), mất nucleotide (-), thêm nucleotide (+)
Dòng mang đột biến đồng hợp tử ở cả hai gen (DT1.1-5, DT1.1-13) tiếp tục
được duy trì ổn định ở thế hệ này (mất đoạn 77 bp ở gen GmGOLS03 và -23 bp ở gen GmGOLS19). Dòng DT1.1-7, DT1.1-14 được sàng lọc để thu được cá thể mang đột biến đồng hợp tử ở gen GmGOLS03 và không mang đột biến ở gen GmGOLS19.
Kết hợp kết quả phân tích sự phân ly và giải trình tự đã thu được dòng DT1.1- 7-1 mang đột biến đồng hợp tử đồng thời ở hai gen (-32 bp ở gen GmGOLS03 và -23 bp ở gen GmGOLS19), dòng DT1.1-7-2 mang đột biến đồng hợp tử chỉ ở gen GmGOLS03 (-32 bp ở gen GmGOLS03) và không mang đột biến ở gen GmGOLS19, và dòng DT1.1-14-3 mang đột biến đồng hợp tử chỉ ở gen GmGOLS03 (-77 bp ở gen GmGOLS03) và không mang đột biến ở gen GmGOLS19.
Như vậy, kết quả phân tích ở hệ T2 đã khẳng định sự di truyền ổn định của các đột biến định hướng của gen GmGOLS trên các dòng đậu tương có nguồn gốc từ giống ĐT26. Chúng tôi đã thu được các dòng đậu tương mang đột biến đồng hợp trên đơn gen hoặc cả hai gen nghiên cứu để phục vụ cho các phân tích về sinh trưởng, phát triển cũng như thành phần hạt ở các nghiên cứu tiếp theo.
3.4.2 Các dòng M3.1 và M4.1 của giống Marverick
3.4.2.1 Sự di truyền của gen chọn lọc bar ở thế hệ T1
Tương tự như giống ĐT26, bằng phương pháp phết thuốc diệt cỏ glufosinate nồng độ 200 mg/l để nhận diện gen bar và sàng lọc PCR sự có mặt của trình tự T- DNA sử dụng cặp mồi đặc hiệu cho gen pcoCas9 và vùng mở rộng 35SPPDK – pFGC trong gen được tiến hành tương tự như thế hệ T1 của dòng DT1.1. Kết quả đã thu được 4 dòng đậu tương đột biến thế hệ T1 (M3.1-2, M3.1-6, M4.1-1, M4.1-5) của giống Mr không phản ứng với glufosinate 200 mg/l sau 5 ngày thực hiện phương pháp phết lá, kết quả ghi nhận tại Bảng phụ lục 2. Thông qua phương pháp điện di sản phẩm PCR của các mẫu T1 thu được với các cặp mồi đặc hiệu (Bảng phụ lục 1) trên gel agarose 1% đã ghi nhận băng vạch xuất hiện rõ nét ở 4 dòng (M3.1-2, M3.1- 6, M4.1-1, M4.1-5) thế hệ T1. Điều này chứng tỏ gen bar di truyền một cách ổn định từ thế hệ cây T0 sang cây T1.
3.4.2.2 Sự di truyền, phân ly của các đột biến gen GmGOLS ở thế hệ T1
Sự di truyền và phân ly của các đột biến tạo ra bởi CRISPR/Cas9 ở thế hệ T1 của các dòng M3.1 và M4.1 thuộc giống Mr cũng được nghiên cứu bằng cách điện di trên gel và giải trình tự (Hình 3.16). Ở thế hệ T0 kết quả ghi nhận từ Hình 3.14 cho thấy rằng các dòng M3.1 và M4.1 đều mang đột biến thể khảm (chimeric) ở hai gen
GmGOLS03 và GmGOLS19. Tuy nhiên, hầu hết các đột biến không được truyền sang thế hệ tiếp theo. Kết quả cụ thể về dạng phân ly và kiểu gen của các dòng đột biến thế hệ T1 được thống kê ở bảng 3.3.
Đối với GmGOLS03, chỉ một số alen nhất định xuất hiện ở cây T0 như dòng mất đoạn Δ-33 bp ở dòng M3.1 và mất đoạn Δ-30 bp của dòng M4.1 được di truyền, trong khi các alen mới cũng được quan sát thấy ở thế hệ T1. Ngoài ra, các alen đột biến mới của gen GmGOLS19 được phát hiện ở thế hệ T1 mà không quan sát thấy ở cây T0. Điều quan trọng là các alen đột biến mới mất đoạn Δ-30 bp xuất hiện ở dạng đồng hợp tử và hay mất đoạn Δ-22 bp và Δ-33 bp xuất hiện ở dạng biallelic ở thế hệ T1.
Bảng 3.3 Danh sách các dòng đậu tương mang đột biến gen GmGOLS03 và
GmGOLS19
Dạng đột biến (*) | Kiểu gen (**) | Dạng phân ly ở thế hệ T2 (GmGOLS03// GmGOLS19) | Kiểu gen ở thế hệ T2 (GmGOLS03// GmGOLS19) | |||
GmGOLS03 | GmGOLS19 | GmGOLS03 | GmGOLS19 | |||
M3.1-2 | -22/-33 | 0 | Biallelic | WT | -22//0 | Homo//WT |
-33//0 | Homo//WT | |||||
-22/-33//0 | Biallelic//WT | |||||
M3.1-6 | -33 | -22 | Homo | Homo | -33/-22 | Homo//Homo |
M3.1-9 | -22/-33 | -22 | Biallelic | Homo | -22//-22 | Homo//Homo |
-33//-22 | Homo//Homo | |||||
-22/-33//-22 | Biallelic//Homo | |||||
M4.1-2 | -30/0 | 0 | Hetero | WT | -30//0 | Homo//WT |
0//0 | WT//WT | |||||
-30/0//0 | Hetero//WT | |||||
M4.1-4 | -30 | 0 | Homo | WT | -30//0 | Homo//WT |
M4.1-5 | -30 | -22 | Homo | Homo | -30//-22 | Homo//Homo |
Có thể bạn quan tâm!
-
 Chuyển Gen Tạo Cây Đậu Tương Mang Cấu Trúc Crispr/cas9
Chuyển Gen Tạo Cây Đậu Tương Mang Cấu Trúc Crispr/cas9 -
 Sơ Đồ Minh Họa Cấu Trúc Vector Chỉnh Sửa Gen Crispr/cas9 Được Thế Kế Để Chuyển Gen Vào Đậu Tương
Sơ Đồ Minh Họa Cấu Trúc Vector Chỉnh Sửa Gen Crispr/cas9 Được Thế Kế Để Chuyển Gen Vào Đậu Tương -
 Kiểm Tra Hoạt Động Của Cấu Trúc Chỉnh Sửa Gen Crispr/cas9 Trên Các Dòng Rễ Tơ
Kiểm Tra Hoạt Động Của Cấu Trúc Chỉnh Sửa Gen Crispr/cas9 Trên Các Dòng Rễ Tơ -
 Kết Quả Phân Tích Sản Phẩm Pcr Của Chỉ Thị Phân Tử Gols-Seg F1 Và Gols-Seg F3 Với Các Cây Đột Biến T2
Kết Quả Phân Tích Sản Phẩm Pcr Của Chỉ Thị Phân Tử Gols-Seg F1 Và Gols-Seg F3 Với Các Cây Đột Biến T2 -
 Tỉ Lệ Carbohydrate Dạng Stachyose Và Sucrose Trên Tổng Khối Lượng Carbohydrate Hòa Tan Trong Hạt Đậu Tương
Tỉ Lệ Carbohydrate Dạng Stachyose Và Sucrose Trên Tổng Khối Lượng Carbohydrate Hòa Tan Trong Hạt Đậu Tương -
 Việc Lựa Chọn Trình Tự Mục Tiêu Trong Thiết Kế Cấu Trúc Chỉnh Sửa Gen Trên Đậu Tương
Việc Lựa Chọn Trình Tự Mục Tiêu Trong Thiết Kế Cấu Trúc Chỉnh Sửa Gen Trên Đậu Tương
Xem toàn bộ 122 trang tài liệu này.
Ghi chú: (*) Dấu “-” thể hiện đột biến mất đoạn. (**) WT thể hiện gen quan tâm không đột biến, “Hetero” thể hiện sự đột biến xảy ra ở một alen của gen trong khi alen còn lại không đột biến, “homo” thể hiện sự đột biến xảy ra đồng thời ở hai alen và đột biến là giống nhau trên hai alen, và “biallelic” thể hiện 2 đột biến khác nhau xảy ra trên hai alen của một gen
3.4.2.3 Sự di truyền, phân ly của đột biến gen GmGOLS ở thế hệ T2
Nhằm rút ngắn và nâng cao hiệu quả của công tác kiểm tra sự di truyền của
các đột biến định hướng, đặc biệt là các đột biến mất đoạn ngắn của gen quan tâm trong các dòng đậu tương ở các thế hệ tiếp theo, chúng tôi tiến hành xây dựng và sử dụng các chỉ thị phân tử là các mồi đặc hiệu với từng dạng đột biến. Sự di truyền của các alen GmGOLS đột biến ở thế hệ T2 từ các dòng T1 được đánh giá bằng các chỉ thị phân tử kết hợp với phương pháp điện di trên gel agarose và giải trình gen. Trong đó, chỉ thị GOLS-seg F được thiết kế đặc hiệu để nhận biết alen WT (tức là alen không đột biến) và alen đột biến mất 22 bp (Δ-22 bp); chỉ thị GOLS-seg F1 được thiết kế để nhận biết alen mang đột biến mất 33 bp (Δ-33 bp)và chỉ thị GOLS-seg F3 được thiết kế để nhận biết alen mang đột biến mất 30 bp (Δ-30 bp). Khi một trong các chỉ thị phân tử kết hợp với một mồi đặc hiệu cho gen GmGOLS03 hoặc GmGOLS19, thông qua phân tích sản phẩm PCR các alen được khuếch đại và xuất hiện băng vạch khi điện di trên gel agarose 2%.
Kết quả sàng lọc của các đột biến thông qua phân tích sản phẩm PCR sẽ thu được như dự kiến ban đầu tại Bảng 2.3.
Kết quả sàng lọc các đột biến bằng các chỉ thị phân tử như sau:
Chỉ thị phân tử GOLS-seq F: GOLS-seg F được thiết kế với mục tiêu kết hợp với mồi đặc hiệu G03R để phát hiện alen đột biến Δ-22 bp của gen GmGOLS03 (Bảng 2.3). Tuy nhiên, với thiết kế này kết quả PCR cũng sẽ khuếch đại được alen không đột biến (WT) của các dòng đậu tương nghiên cứu. Tương tự như vậy, khi kết hợp GOLS-seg F với mồi G19 R, các dòng mang alen đột biến Δ-22 bp và alen WT của gen GmGOLS19 sẽ thu được sản phẩm PCR đặc hiệu. Sử dụng cây T2 của 6 dòng T1 mang các dạng đột biến khác nhau (Bảng 3.3) để tiến hành phân tích với chỉ thị phân tử GOLS-seq F, chúng tôi thu được kết quả trên Hình 3.18. Đối với dòng M3.1-2, các cây T2 được kiểm tra đều cho một băng bạch DNA rõ nét (500 bp) khi sử dụng kết hợp GOLS-seq F cùng mồi đặc hiệu G03 R của gen GmGOLS03 (Hình 3.18A). Điều này chứng tỏ các cây T2 của dòng M3.1-2 mang alen đột biến Δ-22 bp (ở dạng đồng hợp tử -22/-22 bp hoặc bialleilc -22/-33 bp) trên gen GmGOLS03. Trên gen GmGOLS19, sử dụng kết hợp GOLS-seq F cùng mồi đặc hiệu G19 R các cây T2 của dòng này đều có kết quả xuất hiện 1 băng vạch DNA, chứng tỏ sự có mặt của alen WT (Hình 3.18B).
Ở dòng M3.1-6, các cây T2 được kiểm tra đều không có sản phẩm PCR, chứng tỏ sự vắng mặt của alen Δ-22 bp và alen WT trên gen GmGOLS03 (Hình 3.18A).
Trên gen GmGOLS19, các cây T2 này đều xuất hiện băng vạch PCR đặc hiệu, điều này cho thấy sự di truyền của alen đột biến Δ-22 bp dạng đồng hợp tử (Hình 3.18B). Với dòng M3.1-9, các cây T2 đều có sự xuất hiện băng vạch DNA đặc hiệu,
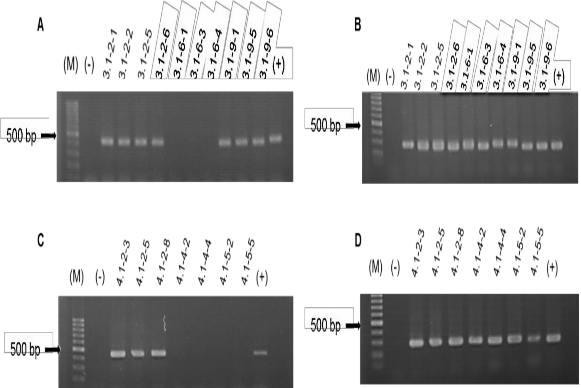
chứng tỏ chúng có mang alen đột biến mất -22 bp trên gen GmGOLS03 (Hình 3.18A). Tuy nhiên, tương tự như ở dòng M3.1-2, chỉ thị phân tử này cũng chưa chỉ ra được kiểu gen đồng hợp tử hoặc hoặc hai alen đột biến của gen GmGOLS03. Trên gen GmGOLS19 các cây T2 được kiểm tra của dòng M3.1-9 đều có kết quả xuất hiện băng vạch thể hiện tính duy trì ổn định của alen đột biến mất Δ-22 bp ở dạng đồng hợp tử.
Hình 3.18. Kết quả phân tích sản phẩm PCR của chỉ thị phân tử với các cây đột biến T2
A, C. Sản phẩm PCR khi kết hợp GOLS-seq F và mồi đặc hiệu G03 R; B,D. Sản phẩm PCR khi kết hợp GOLS-seq F và mồi đặc hiệu G19R; (-). Đối chứng âm sử dụng nước de-ion; (+). Đối chứng dương là DNA dòng cây không đột biến; M. Thang DNA chuẩn 100 bp
Ngoài ra, các cây T2 của dòng M4.1-2 đều có xuất hiện băng vạch PCR đặc hiệu, cho thấy tính di truyền của alen WT của gen GmGOLS03 (Hình 3.18C). Trong khi đó, toàn bộ cây T2 của hai dòng M4.1-4 và M4.1-5 đều không thu được sản phẩm