tại điểm đến | |||||||
24 | HATT2 | Sự thân thiện của người dân địa phương đối với du khách | 1 | 2 | 3 | 4 | 5 |
25 | HATT3 | Điểm đến thú vị, hấp dẫn và thoải mái | 1 | 2 | 3 | 4 | 5 |
26 | HATT4 | Trải nghiệm tổng thể về điểm đến được cung cấp “phù hợp” với nhu cầu của tôi | 1 | 2 | 3 | 4 | 5 |
27 | SAT1 (SAT2 cǜ) | Du lịch ở đây tốt hơn mong đợi | 1 | 2 | 3 | 4 | 5 |
28 | SAT2 (SAT3 cǜ) | Đánh giá chung của tôi về trải nghiệm khi du lịch nơi này là rất tốt | 1 | 2 | 3 | 4 | 5 |
29 | SAT3 (SAT4 cǜ) | Tôi hài lòng với điểm đến này | 1 | 2 | 3 | 4 | 5 |
30 | INT1 | Tôi mong muốn quay trở lại điểm du lịch này | 1 | 2 | 3 | 4 | 5 |
31 | INT2 (INT3 cǜ) | Tôi có ý định quay lại để du lịch vào một thời điểm trong tương lai gần | 1 | 2 | 3 | 4 | 5 |
32 | INT3 (INT4 cǜ) | Tôi sẽ giới thiệu nơi này cho người xung quanh | 1 | 2 | 3 | 4 | 5 |
Có thể bạn quan tâm!
-
 Chuẩn Chủ Quan Của Du Khách Đối Với Điểm Đến
Chuẩn Chủ Quan Của Du Khách Đối Với Điểm Đến -
 Kiểm Soát Hành Vi Của Du Khách Đối Với Điểm Đến
Kiểm Soát Hành Vi Của Du Khách Đối Với Điểm Đến -
 Năm Sinh: ……………………………………………….……………………
Năm Sinh: ……………………………………………….…………………… -
 Các nhân tố ảnh hưởng đến ý định quay lại điểm đến du lịch của du khách: Trường hợp 3 tỉnh ven biển Tây Nam sông Hậu là Cà Mau, Bạc Liêu và Sóc Trăng Việt Nam - 27
Các nhân tố ảnh hưởng đến ý định quay lại điểm đến du lịch của du khách: Trường hợp 3 tỉnh ven biển Tây Nam sông Hậu là Cà Mau, Bạc Liêu và Sóc Trăng Việt Nam - 27
Xem toàn bộ 217 trang tài liệu này.
XIN CẢM ƠN!
PHỤ LỤC 13
PHÂN TÍCH MÔ HÌNH CẤU TRÚC TUYẾN TÍNH SEM TRONG NGHIÊN CỨU CHÍNH THỨC
Model | NPAR | CMIN | DF | P | CMIN/DF |
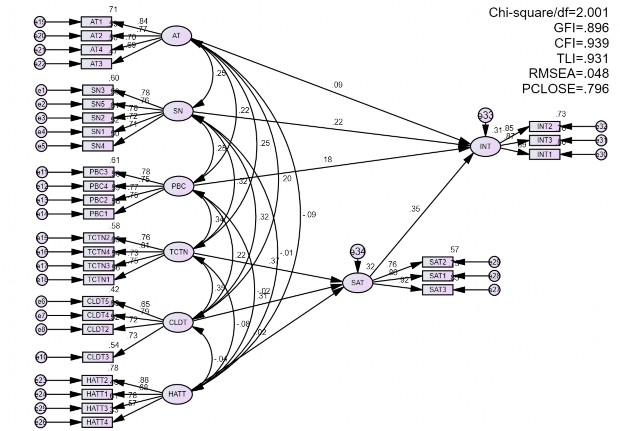
Default model | 84 | 824.497 | 412 | .000 | 2.001 |
Saturated model | 496 | .000 | 0 | ||
Independence model | 31 | 7234.119 | 465 | .000 | 15.557 |
Model Fit Summary CMIN
RMR, GFI
RMR | GFI | AGFI | PGFI | |
Default model | .031 | .896 | .875 | .744 |
Saturated model | .000 | 1.000 | ||
Independence model | .134 | .337 | .293 | .316 |
Baseline Comparisons
NFI Delta1 | RFI rho1 | IFI Delta2 | TLI rho2 | CFI | |
Default model | .886 | .871 | .940 | .931 | .939 |
Saturated model | 1.000 | 1.000 | 1.000 |
NFI Delta1 | RFI rho1 | IFI Delta2 | TLI rho2 | CFI | |
Independence model | .000 | .000 | .000 | .000 | .000 |
Model
Parsimony-Adjusted Measures
PRATIO | PNFI | PCFI | |
Default model | .886 | .785 | .832 |
Saturated model | .000 | .000 | .000 |
Independence model | 1.000 | .000 | .000 |
NCP
NCP | LO 90 | HI 90 | |
Default model | 412.497 | 334.540 | 498.231 |
Saturated model | .000 | .000 | .000 |
Independence model | 6769.119 | 6497.072 | 7047.588 |
FMIN
FMIN | F0 | LO 90 | HI 90 | |
Default model | 1.865 | .933 | .757 | 1.127 |
Saturated model | .000 | .000 | .000 | .000 |
Independence model | 16.367 | 15.315 | 14.699 | 15.945 |
RMSEA
RMSEA | LO 90 | HI 90 | PCLOSE | |
Default model | .048 | .043 | .052 | .796 |
Independence model | .181 | .178 | .185 | .000 |
AIC
AIC | BCC | BIC | CAIC | |
Default model | 992.497 | 1005.609 | 1336.357 | 1420.357 |
Saturated model | 992.000 | 1069.424 | 3022.411 | 3518.411 |
Independence model | 7296.119 | 7300.958 | 7423.020 | 7454.020 |
ECVI
ECVI | LO 90 | HI 90 | MECVI | |
Default model | 2.245 | 2.069 | 2.439 | 2.275 |
Saturated model | 2.244 | 2.244 | 2.244 | 2.420 |
Independence model | 16.507 | 15.892 | 17.137 | 16.518 |
HOELTER
HOELTER HOELTER .05 .01 | |
Default model Independence model | 247 259 32 33 |
Regression Weights: (Group number 1 - Default model)
Estimate | S.E. | C.R. | P | Label | ||
SAT | <--- | TCTN | .487 | .071 | 6.897 | *** |
SAT | <--- | CLDT | .481 | .086 | 5.588 | *** |
SAT | <--- | HATT | -.032 | .063 | -.506 | .613 |
INT | <--- | AT | .089 | .052 | 1.728 | .084 |
INT | <--- | PBC | .209 | .059 | 3.570 | *** |
INT | <--- | SAT | .320 | .044 | 7.284 | *** |
INT | <--- | SN | .257 | .061 | 4.228 | *** |
SN3 | <--- | SN | 1.000 | |||
SN5 | <--- | SN | 1.066 | .067 | 16.004 | *** |
SN2 | <--- | SN | .973 | .059 | 16.354 | *** |
SN1 | <--- | SN | .878 | .058 | 15.065 | *** |
SN4 | <--- | SN | .925 | .062 | 14.797 | *** |
CLDT5 | <--- | CLDT | 1.000 | |||
CLDT4 | <--- | CLDT | 1.323 | .103 | 12.892 | *** |
CLDT2 | <--- | CLDT | 1.155 | .095 | 12.139 | *** |
CLDT3 | <--- | CLDT | 1.187 | .097 | 12.297 | *** |
PBC3 | <--- | PBC | 1.000 | |||
PBC4 | <--- | PBC | .895 | .059 | 15.234 | *** |
PBC2 | <--- | PBC | .992 | .064 | 15.568 | *** |
PBC1 | <--- | PBC | 1.010 | .066 | 15.242 | *** |
TCTN2 | <--- | TCTN | 1.000 | |||
TCTN4 | <--- | TCTN | 1.040 | .065 | 16.048 | *** |
TCTN3 | <--- | TCTN | .991 | .068 | 14.679 | *** |
TCTN1 | <--- | TCTN | 1.070 | .071 | 15.070 | *** |
AT1 | <--- | AT | 1.000 | |||
AT2 | <--- | AT | .918 | .056 | 16.509 | *** |
AT4 | <--- | AT | .849 | .056 | 15.061 | *** |
AT3 | <--- | AT | .784 | .054 | 14.615 | *** |
HATT2 | <--- | HATT | 1.000 | |||
HATT1 | <--- | HATT | .724 | .049 | 14.658 | *** |
HATT3 | <--- | HATT | .925 | .055 | 16.827 | *** |
HATT4 | <--- | HATT | .606 | .050 | 12.059 | *** |
SAT3 | <--- | SAT | 1.000 | |||
SAT1 | <--- | SAT | .917 | .039 | 23.722 | *** |
SAT2 | <--- | SAT | .846 | .043 | 19.485 | *** |
INT1 | <--- | INT | 1.000 | |||
INT3 | <--- | INT | .982 | .040 | 24.429 | *** |
INT2 | <--- | INT | .940 | .040 | 23.525 | *** |
Standardized Regression Weights: (Group number 1 - Default model)
Estimate | |||
SAT | <--- | TCTN | .372 |
SAT | <--- | CLDT | .309 |
SAT | <--- | HATT | -.024 |
INT | <--- | AT | .086 |
INT | <--- | PBC | .181 |
INT | <--- | SAT | .352 |
INT | <--- | SN | .216 |
SN3 | <--- | SN | .776 |
SN5 | <--- | SN | .764 |
SN2 | <--- | SN | .780 |
SN1 | <--- | SN | .722 |
SN4 | <--- | SN | .711 |
CLDT5 | <--- | CLDT | .651 |
CLDT4 | <--- | CLDT | .794 |
CLDT2 | <--- | CLDT | .719 |
CLDT3 | <--- | CLDT | .733 |
PBC3 | <--- | PBC | .782 |
PBC4 | <--- | PBC | .749 |
PBC2 | <--- | PBC | .766 |
PBC1 | <--- | PBC | .749 |
TCTN2 | <--- | TCTN | .758 |
TCTN4 | <--- | TCTN | .805 |
TCTN3 | <--- | TCTN | .732 |
TCTN1 | <--- | TCTN | .751 |
AT1 | <--- | AT | .845 |
AT2 | <--- | AT | .769 |
AT4 | <--- | AT | .705 |
AT3 | <--- | AT | .686 |
HATT2 | <--- | HATT | .883 |
HATT1 | <--- | HATT | .681 |
HATT3 | <--- | HATT | .783 |
HATT4 | <--- | HATT | .574 |
SAT3 | <--- | SAT | .923 |
SAT1 | <--- | SAT | .863 |
SAT2 | <--- | SAT | .756 |
INT1 | <--- | INT | .895 |
INT3 | <--- | INT | .874 |
INT2 | <--- | INT | .852 |
Squared Multiple Correlations: (Group number 1 - Default model)
Estimate | ||
SAT | .317 | |
INT | .313 | |
INT2 | .725 | |
INT3 | .764 | |
INT1 | .801 | |
SAT2 | .572 | |
SAT1 | .745 | |
SAT3 | .852 | |
HATT4 | .329 | |
HATT3 | .614 | |
HATT1 | .464 | |
HATT2 | .779 | |
AT3 | .471 | |
AT4 | .497 | |
AT2 | .592 | |
AT1 | .713 | |
TCTN1 | .564 | |
TCTN3 | .535 | |
TCTN4 | .649 | |
TCTN2 | .575 | |
PBC1 | .561 | |
PBC2 | .587 | |
PBC4 | .561 | |
PBC3 | .611 | |
CLDT3 | .537 | |
CLDT2 | .517 | |
CLDT4 | .630 | |
CLDT5 | .424 | |
SN4 | .505 | |
SN1 | .522 | |
SN2 | .608 | |
SN5 | .583 | |
SN3 | .602 |
PHỤ LỤC 14
PHÂN TÍCH BOOTSTRAP TRONG NGHIÊN CỨU CHÍNH THỨC
Iterations | Method 0 | Method 1 | Method 2 |
1 | 0 | 0 | 0 |
2 | 0 | 0 | 0 |
3 | 0 | 0 | 0 |
4 | 0 | 0 | 0 |
5 | 0 | 0 | 0 |
6 | 0 | 0 | 0 |
7 | 0 | 1 | 3 |
8 | 0 | 112 | 1 |
9 | 0 | 442 | 2 |
10 | 0 | 469 | 0 |
11 | 0 | 275 | 0 |
12 | 0 | 109 | 0 |
13 | 0 | 30 | 0 |
14 | 0 | 16 | 0 |
15 | 0 | 15 | 0 |
16 | 0 | 4 | 0 |
17 | 0 | 4 | 0 |
18 | 0 | 6 | 0 |
19 | 0 | 11 | 0 |
Total | 0 | 1494 | 6 |
Summary of Bootstrap Iterations (Default model) (Default model)
0 bootstrap samples were unused because of a singular covariance matrix. 0 bootstrap samples were unused because a solution was not found.
Estimate | S.E. | C.R. | P | Label | |||
SAT | <--- | TCTN | .487 | .071 | 6.897 | *** | |
SAT | <--- | CLDT | .481 | .086 | 5.588 | *** | |
SAT | <--- | HATT | -.032 | .063 | -.506 | .613 | |
INT | <--- | AT | .089 | .052 | 1.728 | .084 | |
INT | <--- | PBC | .209 | .059 | 3.570 | *** | |
INT | <--- | SAT | .320 | .044 | 7.284 | *** | |
INT | <--- | SN | .257 | .061 | 4.228 | *** | |
SN3 | <--- | SN | 1.000 | ||||
SN5 | <--- | SN | 1.066 | .067 | 16.004 | *** | |
SN2 | <--- | SN | .973 | .059 | 16.354 | *** | |
SN1 | <--- | SN | .878 | .058 | 15.065 | *** | |
SN4 | <--- | SN | .925 | .062 | 14.797 | *** | |
1500 usable bootstrap samples were obtained. Regression Weights: (Group number 1 - Default model)
Estimate | S.E. | C.R. | P | Label | ||
CLDT5 | <--- | CLDT | 1.000 | |||
CLDT4 | <--- | CLDT | 1.323 | .103 | 12.892 | *** |
CLDT2 | <--- | CLDT | 1.155 | .095 | 12.139 | *** |
CLDT3 | <--- | CLDT | 1.187 | .097 | 12.297 | *** |
PBC3 | <--- | PBC | 1.000 | |||
PBC4 | <--- | PBC | .895 | .059 | 15.234 | *** |
PBC2 | <--- | PBC | .992 | .064 | 15.568 | *** |
PBC1 | <--- | PBC | 1.010 | .066 | 15.242 | *** |
TCTN2 | <--- | TCTN | 1.000 | |||
TCTN4 | <--- | TCTN | 1.040 | .065 | 16.048 | *** |
TCTN3 | <--- | TCTN | .991 | .068 | 14.679 | *** |
TCTN1 | <--- | TCTN | 1.070 | .071 | 15.070 | *** |
AT1 | <--- | AT | 1.000 | |||
AT2 | <--- | AT | .918 | .056 | 16.509 | *** |
AT4 | <--- | AT | .849 | .056 | 15.061 | *** |
AT3 | <--- | AT | .784 | .054 | 14.615 | *** |
HATT2 | <--- | HATT | 1.000 | |||
HATT1 | <--- | HATT | .724 | .049 | 14.658 | *** |
HATT3 | <--- | HATT | .925 | .055 | 16.827 | *** |
HATT4 | <--- | HATT | .606 | .050 | 12.059 | *** |
SAT3 | <--- | SAT | 1.000 | |||
SAT1 | <--- | SAT | .917 | .039 | 23.722 | *** |
SAT2 | <--- | SAT | .846 | .043 | 19.485 | *** |
INT1 | <--- | INT | 1.000 | |||
INT3 | <--- | INT | .982 | .040 | 24.429 | *** |
INT2 | <--- | INT | .940 | .040 | 23.525 | *** |